医学图像人工智能综述
Artificial intelligence as the next step towards precision pathology (SCI 2区,2020)人工智能是精密病理学的下一步
摘要
病理学是癌症护理的基石。个性化癌症治疗需要准确进行生物标志物评估,加大了对癌症组织病理学诊断中的准确率需求的增加。数字图像分析的出现有望改善组织形态学评估的数量和准确性。最近,机器学习,特别是深度学习促进了计算病理学的快速发展。将深度学习整合到日常护理将是下一个十年的护理领域的基石,组织病理学是是这场变革的中心。在病理学中,潜在高价值机器学习应用的例子包括基于日常诊断特征的模型评估与新特征提取和识别的能力,该新特征有望为疾病提供一个新的视角。最近的突破证明了在病理学中,机器学习应用方法极大提高了淋巴结点中癌症的检测效果,乳腺癌中ki67评分,腺癌中Gleason分级和黑色素瘤的肿瘤浸润淋巴细胞(TIL)评分。进一步地,基于深度学习模型已经被证明能够预测标准HE切片的肺、腺、胃和小分子癌症的小分子标志物的状态。此外,在很多疾病中如肺癌、小分子和胃癌,基于HE切片的预后(生存结果)深度神经网络模型已经证明了有效性。在本篇综述中,作者旨在提出和总结在数字图像分析和诊断病理学人工之智能的最新进展。
正文
可以在不损害生物标志物表达的图像分析的情况下,对大型数字化的完整载玻片进行主要压缩和缩放[69]。作者分别介绍了深度学习在各种常见癌症诊断中的最新研究进展。
限制和未来方向
尽管在很多任务中AI目前的结果显示出其能与人类专家性能相匹配的令人信服的结果,AI仍然存在限制和一些挑战。构成深度学习算法临床应用障碍的主要原因之一是解释和理解复杂AI模型如何做出决策的挑战,有时也被称为“黑盒”问题。可解释性AI与可解释性机器学习方法是目前非常活跃的研究领域,提供深度学习模型不同层度上的可解释性解决方案正在出现,可解释性问题在将来可能会减轻,至少部分是这样。建立可解释性机器学习方法对于临床实践而言非常关键,这将会解决一些医学社区常见的批评。另一方面,一旦AI满足所有临床使用,医学参与者同样需要接受一些这种限制。最近的研究表明,当仅用少量数据集进行训练时,目前的AI模型在独立数据集测试阶段将会出现20%的性能下降。
训练数据集的表示误差,如疾病的形态学差异等。关于技术差异的挑战可以通过标准化或严格控制处理减少,如预处理图像数据来减少技术差异的影响,或者通过使模型面对技术差异更加鲁棒来解决。在大量且多样数据集中训练或者fine-tuning深度学习模型有望减少泛化误差。
任何新测试的临床实践的应用都将由规则指导。对于新的AI工具,在CE-IVD认证下非常重要,它可以避免特定实验室机器学习方法的潜在不可复现的风险。定义AI模型必须达到的最低性能水平,以使病理学家们能够接受使用它们,是一个尚未解决的问题。( defining the minimal level of performance that AI models would have to achieve for pathologists to accept using them is an issue that has not been addressed yet.)
结论
随着个性化癌症治疗需求的增加,我们需要更加准确的生物标志物评估和更加定量的组织病理学诊断来治疗并且提高治理决策。病理学家需要配备新的方法论和工具,以满足所需的诊断敏感性和特异性,现在看来,人工智能是迈向精确病理学的下一步。
词汇
- cancer care 癌症护理
- histopathologic 组织病理学
- histomorphological 组织形态学
- holds promise to 有望, be excepted to
- tumour-infiltrating lymphocyte (TIL) 肿瘤浸润淋巴细胞
- gastric 胃的
- prognostic 预后的
- anatomic pathology 解剖病理学
- stromal tissues 基质组织
- be bound to 必然、必定
- reproducibility 再现性、重复能力
- controversy 争论、辩论
- proliferation 增殖,分芽繁殖
- active contour models 主动轮廓模型
- high-through-put 高通量
- assay 化验,分析试验,试样,试料
- equivocal 模棱两可的,意义不明的,模糊的
- inter-observer 观察员之间
- intra-observer 观察者内部
- profile 侧面,轮廓,外形,剖面
- technical variability 技术差异
- stratify 分层
ImageNet Large Scale Visual Recognition Challenge. (2015) ImageNet大规模视觉识别挑战赛(是介绍ImageNet数据集以及其中相应技术)
摘要
ImageNet大规模视觉识别挑战赛是对数百种物体类别和数百万张图像进行物体类别分类和检测的基准。从2010年至今,每年都要进行一次挑战,吸引了50多个机构的参与。
本文描述了该基准数据集的创建以及由此带来的对象识别方面的进步。讨论了收集大规模真实标注数据的挑战,指出了在类别物体识别中的关键突破,提供了当前大规模数据分类和物体检测领域详细分析,并且用人类标准比较了最先进计算机视觉的识别准确率。最后以挑战赛的5年中获得的经验教训作为结束语,并提出未来的方向和改进建议。
An End-to-End Deep Learning Histochemical Scoring System for Breast Cancer TMA (2018, Transaction on medical imaging, TMI, SCI 1区,医学顶刊) 一个乳腺癌TMA的端到端深度学习组织化学评分系统
摘要
诺丁汉预后指数plus一种用于对乳腺癌不同分子类别分层的方法, 该指数使用了与乳腺癌相关的生物标志物对组织微阵列上制备的肿癌组织进行染色。为了决定肿瘤的亚类别,病理学家们不得不通过显微镜对核活性生物标志物进行手工标记,并且使用一个半定量评估方法对每个TMA核心指定一个组织化学得分(H-Score)。手工标记正染色核是一项耗时、不精确、主观过程,这将会导致观察者间与观察者内差异。在本文,作者提出了一种端到端的深度学习系统,它可以直接自动地预测H-Score。系统模仿了病理学家决策过程,使用一个全卷积网络(FCN)来提取肿瘤核区域;一个多列卷积神经网络接收前两个FCN的输出和染色强度描述图像作为输入,作为一个高层决策制定机制来直接得到输入TMA图像的H-Score。就目前而言,该系统是一个使用TMA图像作为输入,并且直接输出临床得分的端到端系统。实验结果证明了使用作者提出的模型能以很高的、与病理学家评分具有显著的统计相关性来预测H-Score,提出算法和病理学家之间H-Score的差异与病理学家之间的主观差异相当。
正文
乳腺癌是一组具有不同基因型和表型特征的异质性肿瘤。最近基因表达谱的研究表明乳腺癌可以分成不同的分子肿瘤组。个性化BC管理通常使用鲁棒的技术,如针对肿瘤分子谱的免疫组织化学(IHC,免疫组化)。为了分类正染色像素和它们的强度,如颜色反卷积的方法在RGB图像中运用数学转换。这些方法被广泛地应用于从负染色中分离出正染色。
对于病理学家和CAD系统而言,传统评估方法至少有3个未解决的问题。首先,正染色强度需要分类成四个类别:未染色、弱、中性和强。然而,分类DAB染色强度尚未有标准定量准则。第一,两个病理学家容易将相同的染色强度分类成两个不同的类别或者将两个不同的强度分类成相同的强度。此外,人类视觉系统可能更多地关注强染色区域,但是这些强染色区域通常围绕着多种染色强度,这将会影响评估结果。第二,在评估中,细胞/核实例计数是一个非常重要的参数。尽管如此,人类和计算机仍然无法很好处理重叠细胞计数的困难。此外,不同核类型的外表的多样性,异质染色和复杂组织结构使得独立分割细胞/核成为一个非常有挑战的问题。第三,肿瘤核和正常核之间外表尺寸的差异将会影响肿瘤核评估的定量判断。在本文,作者想回答这样一个问题:开发一个像有经验的病理学家给出H-Score一样,直接对一个数字病理学图像给出高层评估的CAD模型是否可能?作者的创新在于模仿病理学家做出H-Score的过程,但是却不需要统计几种核的数量。
方法
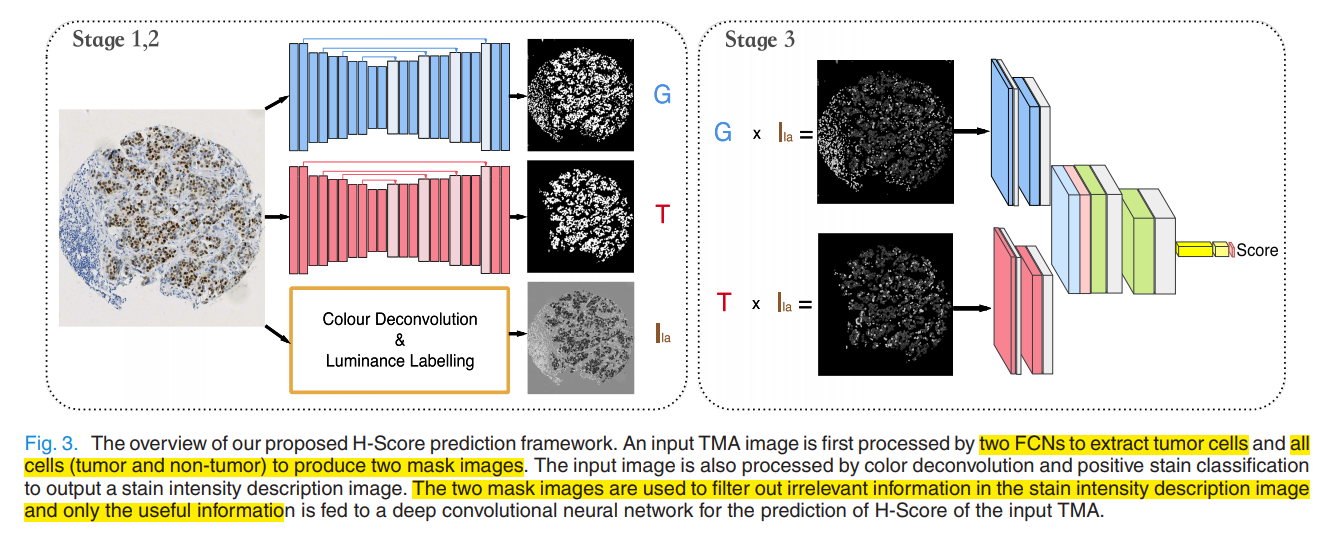
提出的H-Score预测框架包括3个阶段:1)普通核和肿瘤区域分割;2)染色强度描述;3)SINI和SITI的构建,并且通过区域注意力多列卷积神经网络(Region Attention Multi-column Convolutional Neural Network, RAM-CNN)来预测最终的H-Score。框架结构的基本原理是:因为核的数量,肿瘤核的数量和肿瘤核的染色强度个数是预测H-Score的有用信息。因此,作者提出包含这些信息的两个区域掩码。与对染色强度分类人工分界设置不同,作者保持了染色强度的连续描述。仅将有益于预测H-Score的信息提供给深度CNN来预测输入图像的H-Score。这与很多文献中直接将整个图像丢入CNN,而不管区域是否有用的工作不同。
对于正染色像素,为了保留正核的形态学,$I_{la}(m,n)$与原始图像的亮度组件相同;对于负染色像素,在强染色像素(黑蓝颜色)赋予更高的$I_{la}(m,n)$值,赋予更弱染色像素(更亮颜色)更低的值。为了清晰地分离正负像素,往负染色像素中添加了255的偏置,正负像素值将会很清晰地被$I_{la}(m,n)$分离。当$I_{la}(m,n)\leq 255$,它是正染色像素。
使用两个FCN来提取核数量和肿瘤核数量,一个FCN用于分割包括所有核的前景,另一个用于分割仅有肿瘤核的区域。为了分割肿瘤区域,作者对肿瘤TMA图像使用了手工设计的像素层级标签来训练FCN;迁移学习策略被用于普通核检测,训练数据从三个不同的数据集中获取。具体的网络框架如下图所示
实验
由于医学数据集是有限的,因此分别使用了标签和图像增强技术来进行数据增强。其中,为解决模棱两可的H-Score问题,引入了Distributed Label Augmentation(分布式标签增强技术,DLA),即一个实例可能有多个标签。UNET在肿瘤分割中取得了最好的成绩,Res18-UNET在普通核分割中取得最好性能。
在提出的H-Score数据集中,核掩模图像显示使用迁移学习在混合数据集上训练的深度卷积网络可以成功地检测核。肿瘤分割网络能够从正常组织中识别肿瘤区域。值得一提的是,两个检测网络使用了不同的真实掩膜,在Warwick colon癌症图像中所有的核使用了统一的圆形掩膜,肿瘤区域则使用像素级别的标注。因此,由两个网络对相同核生成的最终预测图是不同的。此外,发现随着DAB染色强度的增强,掩膜的扩张变得明显。一个可能的解释是强同质染色使得核纹理核边缘特征变得很难提取。
讨论
不同生物标志物的性能不同,其原因可能是标志物的组织染色不同。尽管这些差异不大,训练一个网络来对不同生物标志物分类将是有益的,并且值得未来研究。
图像越好(TMAs中组织的染色清晰,细胞结构清晰没有严重的重叠),算法的性能就越好。反之,图像越差,结果就越差,也容易被提出的方法误打分。
TMA核中包含大量的失去焦点的区域,这些可能在强染色组织更常发生。模糊区域直接影响核分割的性能和检测准确性。最终它们也影响了回归网络提取拓扑和形态信息。
组织折叠会在薄的组织切片自身折叠时发生,并且在制备载玻片的过程中尤其是在TMA载玻片中很容易发生。 组织折叠会导致幻灯片扫描过程中无法聚焦。 此外,浅色图像中的组织折叠在外观上与深色图像中的肿瘤区域相似[44]。 因此,颜色反卷积的分割精度将在组织折叠区域中受到很大影响。
同质化和重叠同样会影响自动评分的性能。染色同质化会导致在单个核中染色强度差异,并且核重叠会增加其难度。
上述三大挑战将会影响方法的预测,目前很多方法都或多或少存在这些特点中的一两个。因此,未来的工作要克服这些组织来获得更高的预测性能。使用带有先验知识的多种模型的图像预处理是一个方向。
词汇
- histochemical 组织化学
- microarray 微阵列
- nuclei activity 核活性
- semi-quantitative 半定量
- positively stained nuclei 正染色核
- on par with 与。。。相当
- enotype 基因型
- phenotype 表型
- Gene Expression Profiling (GEP)基因表达谱
- antigens 抗原
- Clinical decision making 临床决策
- available treatment options 可用的治疗方案
- Diaminobenzidine (DAB) 二氨基联苯胺(DAB)
- Human Epidermal Growth Factor Receptor 2 (HER2) 人表皮生长因子受体
- rationale 基本原理
- one possible reason is that
Sparse Autoencoder for Unsupervised Nucleus Detection and Representation in Histopathology Images (2019, Pattern Recognition,SCI1区)稀疏编码器,用于组织病理学图像中核检测核表示
摘要
作者提出了一个稀疏卷积编码器(CAE)对组织病理学组织图像进行核检测和特征提取。CAE在组织图像的图像块中检测核和并将核编码成稀疏特征图,该特征图对核的位置与外观进行编码。主要贡献在于通过利用组织病理学图像块特征,提出了一个无监督检测网络。CAE中的预训练核检测核特征提取模块被微调到以端到端方式的监督学习。作者在四个数据集上进行评估并且取得了SOTA的结果。能够以5%的代价取得全监督标注相当的性能。
正文
人们普遍认为,了解和治愈疾病需要对多种生物学规模的疾病机制进行系统的检查,并且需要整合来自多种数据模式的信息。组织样本被长期用于检查在不同子细胞层级和修改组织形态学的疾病自身的表现。很多SOTA核分析方法都是半监督方法,通过预训练一个自编码器学习。无监督表示并且从预训练的自编码器中构建一个CNN。
方法
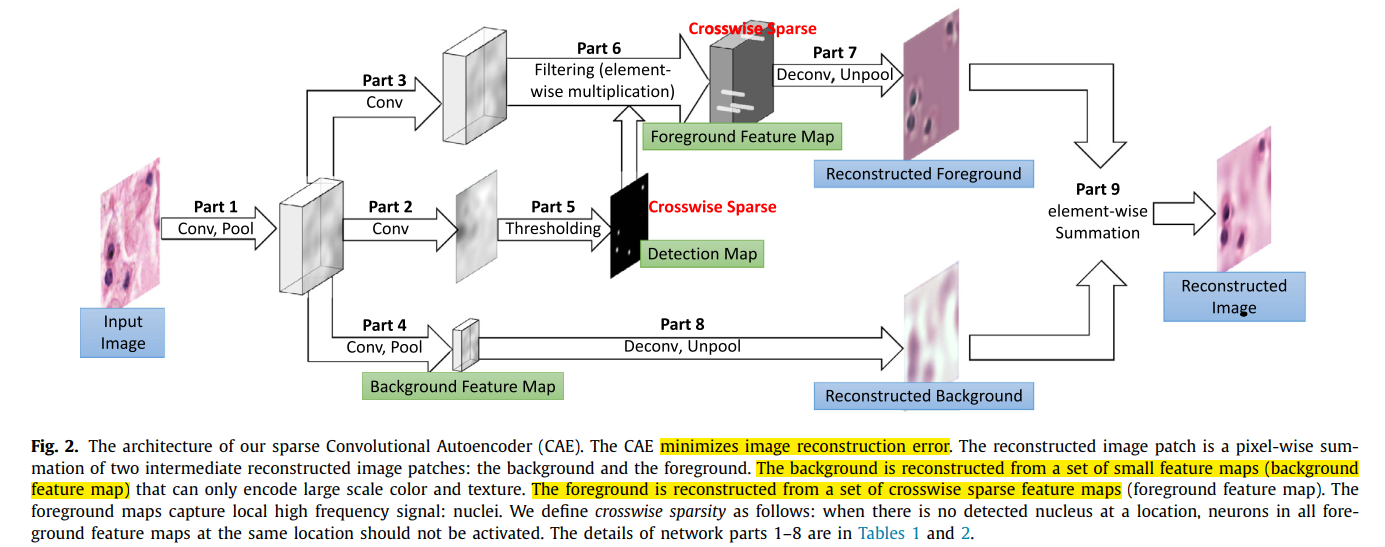
方法修改了传统的CAE,使其不仅对特征图中的外观,还对空域信息进行编码。首先学习分割图像块中的背景(细胞质)和背景(核)。使用图像块的原因是组织图像非常大,内存无法加载。在组织图像分析中将组织图像分成patch并处理是常见的操作。CAE使用少量编码神经元将输入图像编码成低分辨率特征图的集合。受到能力的限制,特征图仅能编码大规模颜色和纹理变化。因此,特征图对图像背景进行编码。
使用滑动窗口方法来训练和测试。CNN输出特征图是输入大小的1/4。使用双线性插值将输出图调整到输入图像大小。最终使用高斯滤波,非极大值抑制和阈值来获得最终的检测的原子核位置。当位于真实原子核周围6个像素内,原子核才算检测正确。如果在真实位置6个像素内有多个位置被检测到,仅将最近的检测位置作为正确位置,其他检测被当作False Positives。
玻片染色的几种方式:Hemeatoxylin and Eosin(H&E), immunohistochemistry (IHC), DAPI 和影像。DAPI染色组织样本中组织图片显示了最高的背景和原子核的对比度。
作者提出的CAE框架如下图所示:
结论
作者提出了一种横向稀疏CAE模型,利用核的视觉特征来同时进行无监督核检测和特征提取。使用CAE初始化监督的CNN,可以以端到端的方式进行核检测,特征提取和分类/分割训练步骤。实验结果显示该方法在效率方面较其他方法好,并且横向约束在提升性能的过程中扮演着一个非常重要的角色。此外,方法可以用其他方法需要的训练数据5%取得与其相当的结果。也调查了使用从DAPI染色图像中生成的弱标签数据集进行训练,实验结果显示可以达到较好的结果。在数字病理图像中生成真实数据是一项费力的过程。这会限制深度学习方法的应用。横向稀疏CAE和弱标签数据解决了数字病理学中的这个问题,并且促进深度学习更有效的应用。在未来的工作中,使用邻域知识来正则编码层,使用如N-CUT技术和更好检测不同形状和纹理的核。对于核实例层级的监督学习分割,将测试深度分水岭方法。为加快测试速度,研究如SqueezeNet网络来减少网络的大小。输入图像与重建背景之间的高频残差是包含核的前景。作者以“横向稀疏”的方式设计网络学习前景特征图:在大多数特征图位置,跨所有特征图的神经元被激活(输出0)。因为未被激活的神经元对后续编码层没有影响,仅使用前景编码特征图中的非0相应来重建前景。这意味着仅当在不同位置捕获到被检测的核的编码神经元被激活,图像重建错误才会最小化。
为了表示参考的核位置,介绍了一种特殊的二值特征图:核检测图。通过二值化激活函数来使这个图变得稀疏。优化以后,当且仅当在核位置中检测到核,在核检测图中的神经元才会输出1。
一旦背景完成编码和重建,重建的背景与输入图像之间的残差就是前景。前景由原子核组成,原子核的大小大致相同,并且通常散布在整个图像。前景编码特征图编码关于原子核的一切,包括位置和外观。前景特征图可以视为一个矩阵,每个条目都是一个向量(神经元响应集合)并编码图像块(原子核的感受域)。如果在图像块中心有原子核,那么向量将编码原子核。否则向量全为0。因为仅使用少量的非0向量编码原子核,所以前景特征图是稀疏的。
词汇
- simultaneous 同时
- deep watershed 深度分水岭方法
- cytoplasm 细胞质
- It is widely accepted that 人们普遍认为
- manifest 表现
- epithelial 上皮
- entry 矩阵中的元素
Structured crowdsourcing enables convolutional segmentation of histology images (2019, SCI 3区)结构化的众包可对组织学图像进行卷积分割(医学图像标注工具)
摘要
动机:在语义图像分割任务中,深度学习算法表现出其突出的性能,构建准确的模型需要大规模标注数据集。由于仔细地描述组织结构需要大量的努力和经验,以及与共享和标记整个切片图像相关存在困难,因此病理学图像的标记是一项挑战。
结果:作者招募了25个参与者,包括有经验的高级病理学家与医学学生,根据数字切片档案对151个乳腺癌切片中组织区域进行标注。系统的评估了参与者之间的不和谐标注,揭露了肿瘤和基质较低的不一致性,对于主观定义的或稀有的组织类别,较高的不一致程度。由高级病理学家提供的反馈实现了2万多个带注释的组织区域的生成和管理。使用这些带注释图像训练的全卷积网络能达到很高的准确率(mean AUC=0.945),并且标注数据的规模显著提高了图像分类的准确率。
正文
病理学图像中组织区域的准确分割是一项具有挑战性的问题,在计算机病理学应用中非常重要。生成有意义的、大规模数量的标注需要很多专家的参与,甚至有经验病理学家也显示出他们之间的不一致性。用于查看和注释全幻灯片组织学图像,协作查看和数据管理的界面也是吸引病理学家扩大准确的地面真相产量的关键要素。在生命科学中,基于游戏化或小额支付的基于人群的方法成功地扩展了生物注释。最近的一项系统综述发现,病理学文献中几乎所有文章都侧重于疟疾诊断和免疫组化生物标记物相对简单的评分。选择ROI代表每个切片中的主导区域类别和纹理。
讨论
卷积网络在分析病理学图像中的成功,引起了研究者们对生成标注数据策略的兴趣。ALs在很多应用中显示出很强大的诊断性能,需要用大规模标注来开发和验证这些模型。在标注研究中,必须有很多参与者,并广泛有效地组织有经验水平的参与者的能力,是一个促进标准过程的方法。病理学的准确分割标注需要非常专业的知识,本文提供了一个例子,即非专家可以训练用于有效地执行这些耗时地工作。但是无法期望非专家可以识别稀有模式或准确标注困难的例子,在勾画组织边界的大部分工作都不属于这个类别。通过在需要的时候利用专业知识,如标记稀有或困难的例子,并且回归或纠正非专业的标注。应当指出的是,许多不一致性是由SP在校正过程中添加的非优势类引起的。
尽管我们的研究提出了有关注释组织学图像的重要发现,但仍有许多问题尚未解决。作者的研究依靠医学学生和应届毕业生,基本原理利用熟悉病理学或普通生成物的人来减少可能错误的打分。未来的研究可能研究这个假设是否正确,以及是否有可能让缺乏此项培训的更多参与者参与进来,以进一步扩大注释工作的规模。作者的研究没有评估参与者内的不和谐性,这是病理学中众所周知非常重要的一个问题。
词汇
- crowdsourcing 众包
- delineate 勾画
- in the context of 在。。。背景下
- to a lesser extent 在较小的范围下
- granular 粒状
- asymptotic 渐进趋势
Learning Where to See: A Novel Attention Model for Automated Immunohistochemical Scoring(2019, TMI, SCI1区)学习在哪里看:自动化免疫组织化学评分的新型注意模型
摘要
估算入侵乳腺癌中HER2的过度放大的范围被认为是很重要的预测和诊断标记。作者提出一种新颖的基于深度强化学习(DRL)模型,将HER2的免疫组织化学(IHC)评分作为一个序列学习任务。对于从一个从多分辨率大尺寸全切片图像(WSI)中给定图像图块采样,该模型学习通过遵循参数化策略来顺序地识别一些诊断相关的兴趣区域(ROI)。通过循环和残差卷积网络处理选择的ROI,学习为不同HER2评分和预测下一个位置的判别性特征,而不需要处理所有给定图像图块的亚图像块来预测HER2得分,模仿组织病理学家不需要以最高放大倍数分析切片的每个部分的过程。提出的模型合并了一个特定任务正则化项和抑制返回机制来防止模型访问之前注意的位置。作者在两个IHC数据集上进行了评估:一个HER2评分挑战比赛的公开可用的数据集和其他由Glo1标记染色的胃肠胰腺神经内分泌肿瘤切片组成的WSI图像数据集。证明了提出模型优于其他基于深度卷积网络的SOTA方法。就作者所知,这是第一项使用DRL来进行IHC评分并且潜在地促进DRL在计算病理学领域地广泛使用的研究,该研究可以减少大规模千兆像素病理学图像分析的计算负担。
正文
在具有侵袭性肿瘤区域的乳腺癌(BC)病例中观察到HER2基因的过度扩增。与HER2-(阴性)病例相比,BC HER2 +(阳性)病例的癌细胞会发生肿瘤转化,导致肿瘤细胞不受控制的生长。 推荐将所有浸润性乳腺癌病例用于HER2检测[1],近20-30%的病例HER2蛋白过表达[2],这与预后不良,生存率较低和复发率高度相关。最近的研究报道了HER2的状态是抗HER2和激素治疗的预测因素,也是将浸润性肿瘤与死亡率和无复发生存时间相关的预后因素。因此,准确定量HER2过表达对于确保HER2 +患者接受适当的抗HER2治疗至关重要。
IHC染色是HER2生物标志物表达最常见的方法。在常规临床实践中,专业病理学家在显微镜下目视检查IHC染色的BC组织学切片,以定量HER2的表达,并报告0到3+之间的得分。得分为0或1+的样品在不到10%的肿瘤细胞中没有膜染色或膜染色很弱,被视为HER2-。 在超过10%的肿瘤细胞中观察到强烈的膜染色并被视为HER2 +的情况,得分为3+。 观察到染色不均匀的临界病例被分类为模棱两可,得分为2+。据报道,超过20%的HER2结果可能都不正确。
对于手头的任务,通常在整个WSI基本上提供IHC分数的真实标签,并且没有提供关于做出最终HER2得分的组织切片的ROIs的详细标注。在现有的自动标注方法中,最简单的方法是手工或随机从WSI中提取ROIs块来训练一个监督模型来预测所需的HER得分。这些方法不可避免地对模型预测有偏差并且块的不连续选择也可能会遭受视觉环境的损失。其他潜在的缺陷是计算效率较低,因为这些方法需要处理给定图像中所有的区域,这些区域中的一些组织区域可能跟预测正确的HER2得分的诊断不相关。
关于上述提到的挑战,作者提出这样的问题:能够训练一个忽略不相干信息,学习“看哪里”的模型?文中提出的模型通过学习ROI位置相互作用序列中的参数化策略,从给定图像的低分辨率(2.5倍)粗粒度表示中识别出一些与诊断相关的位置。该模型从相关位置顺序采样40x和20x的多分辨率ROI,以学习针对不同HER2分数(0至3+)的判别特征。该模型的核心组件是残差卷积神经网络(ResNet)和递归神经网络(RNN)。 ResNet在此模型中的作用是学习判别功能,而RNN依次分析提供的功能以预测结果和下一个位置。 由于GT信息仅提供给WSI级别,而没有ROI位置的先验知识,因此我们使用策略梯度来训练模型。 我们的模型旨在探索空间上不同的位置,并从视觉上有区别的区域中学习特征。 在认知心理学中,这种现象称为回返抑制(IoR)[8],它阻止了以前参加的区域再次参加。 我们的模型结合了IoR的概念,以鼓励模型参与非重叠的诊断相关区域。为减少错误得将3+得分设置为0/1+,提出了一个特定于任务的正则化项,它会惩罚此类预测。
最常见的自动IHC评分方法包括一个预处理步骤来识别潜在组织区域来训练潜在的模型。然后,手动地从选择地组织区域中采样一小块,通过随机或使用滑动窗口的方法。识别潜在组织区域通常有三种方法:手工选择,基于半自动化或阈值的方法。必须探索如何训练深度学习模型以忽略给定图像中的不必要信息,而仅关注有助于最终预测正确结果的区域。另一个有趣的扩展是将注意模型与策略梯度相结合,使下划线模型能够学习基于空间依赖性的参数化策略。
模型最重要的任务预测下一个$l_{t+1}$的位置,根据由CNN提供的$v^{\downarrow16}$以及由CNN和LSTM处理ROI图像的隐藏层表示$f_h(\theta_h)$。通过计算$v^{\downarrow16}$和$f_h(\theta_h)$之间的Hadamard乘积来获得组合特征向量。最终定位模型$f_l(\theta_l)$,线性转换组合特征来预测下一个位置$l_{t+1}(x,y)$的归一化坐标。
网络结构如图所示
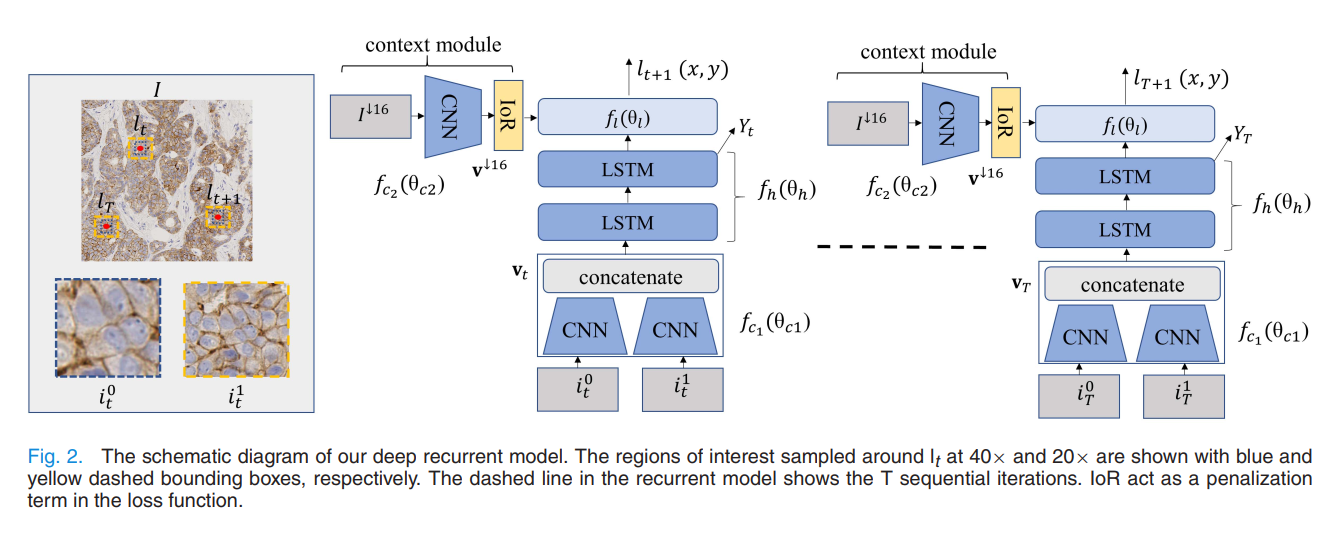
从图中可以看出由四个模型构成:三个卷积网络和一个循环神经网络。其中context module模块网络用于处理输入的组织patch图像,即$f_{c2}(\theta_{c2})$。另外$f_{c1}(\theta_{c1})$用于提取组织Patch图像中ROI块的40x和20x的特征(分辨率不同如何输入同一个网络?),之后在将两个特征合并。RNN则是接收$f_{c1}(\theta_{c1})$的输出。最终模型$f_l(\theta_l)$接收RNN模型的输出和$f_{c2}(\theta_{c2})$的输出,来预测下一个ROI的位置。模型的任务是学习参数化DRL策略$(\pi)$,该策略遵循参数化策略$(\pi)$最大化期望奖励将给定状态映射到动作。
在诊断相关的区域具有充分选择的一个重要因素是抑制以前访问区域的模型。这种简单的抑制回报(IoR)策略导致该模型将更高的优先级赋予该区域,而该区域先前并未被用来学习判别特征。为实现该目的,提出$L_{IoR}$损失项来计算选择的ROIs之间的重叠坐标,并惩罚选择彼此之间相当较近的图像块。
通常,WSI的大部分包含没有组织成分的背景(玻璃)区域。 对于组织分割,我们在WSI的较低分辨率(2.5倍)版本上执行局部熵过滤。
现有的基于注意力的模型[23],[36],包括RMVA(视觉注意力的递归模型),都没有机制来阻止模型重新访问先前参与的区域。 此外,RMVA还具有在模型之前的强大位置,这在组织学图像分析中是无关紧要的,这主要是由于随机定向和基础组织的形态外观所致。最具挑战的图像来自HER2中的类别1+和2+,这些区域由于肿瘤在形态病理学表现呈现出同质化的特点。
使用8个ROI时性能相对较低的主要原因之一是包含组织边界区域的图像,其中大部分图像区域被背景玻璃覆盖。 因此,在那些情况下,选择更多的位置可能会使模型在预测正确的结果时感到困惑。以更高的分辨率选择的ROI在细胞水平上提供了有关HER2表达的更详细的信息。 另一方面,以10倍或更低放大倍数选择的ROI提供了更多的上下文信息,但它们也更有可能包含背景或无关的问题区域,包括非侵入性苏木染色区域。
在HER2评分中,限制ROI的尺寸来防止包含不相关组织区域是很重要的。其他预后和预测指标如雌激素受体(ER),孕激素受体(PR)和Ki-67在WSI水平上的增殖也可以通过类似方法评分。
结论
本文提出了一种深度强化学习的方法来自动对IHC染色的乳腺癌的HER2切片评分。不同于处理给定输入图像所有区域的全监督模型,提出模型将IHC评分视为一个序列选择任务,并且可以有效地定位诊断相关的区域(where to see)。提出模型可以潜在地解决其他准确像素级别标注的病理学图像分析问题。相似的方法也许也可以辅助病理学家对在H&E和IHC染色病理学图像中潜在ROI的自动定位和分类。在公开可用的HER2评分挑战数据集上评估。详细的比较验证支撑了提出的模型。这项研究可能会开始在计算病理学领域将顺序学习和策略梯度进行更可解释的整合。
词汇
- estimating 估算
- human epidermal growth factor receptor 2(HER2) 人类表皮生长因子受体2
- over-amplification 过度放大
- inhibition 抑制,禁忌,禁令
- gastroenteropancreatic 胃肠胰
- neuroendocrine 神经内分泌
- poor prognosis 预后不良
- underlying model 基础模型
- finite horizon problem 有限范围问题
- imperative 至关重要的
- ineludible 不可避免的
Pixel-to-Pixel Learning With Weak Supervision for Single-Stage Nucleus Recognition in Ki67 Images (2019, SCI2区, TBME)对Ki67图像中的单阶段核识别进行弱监督的像素间学习。(一步到位,直接以端到端的方式预测,不需要分割过程)
摘要
对象:核识别在组织病理学分析中是一项关键的挑战,如在Ki67免疫组织化学染色图像中。尽管已经有很多标注方法提出,但大多数使用多阶段处理管道来分类核,非常麻烦。低通量核的评估容易出错。为解决该问题,作者提出一种新颖的全卷积网络来进行单阶段核识别。
方法:与直接对像素层面分类不同,文章构建了核识别作为一个深度结构化的回归模型。对每张输入图像,它输出多个近似图,每个近似图与一个核类别相对应,并且在核中心区域表现出强烈的响应。此外,考虑到在组织病理学图像中核分布,我们进一步介绍了一个辅助任务,ROI的提取,用弱ROI来协助并促进核的量化。提出的网络以端到端、像素间的方式学习模拟核检测与分类。结果:在胰神经内分泌肿瘤Ki67图像数据集上的验证网络的性能,实验证明文章的方法优于最近SOTA方法。结论:提出了一种新的,像素间的双分支深度神经网络来有效地识别核,并与其他相关任务一同学习,如ROI提取,可以进一步促进单个核定位和分类。重要性:作者的方法提供了一个干净,单阶段核识别管道对组织病理学图像分析,特别是为Ki67图像定量化提供了一个新的方向,潜在地益于全切片图像中单目标的量化。
作者提出的方法如下图所示
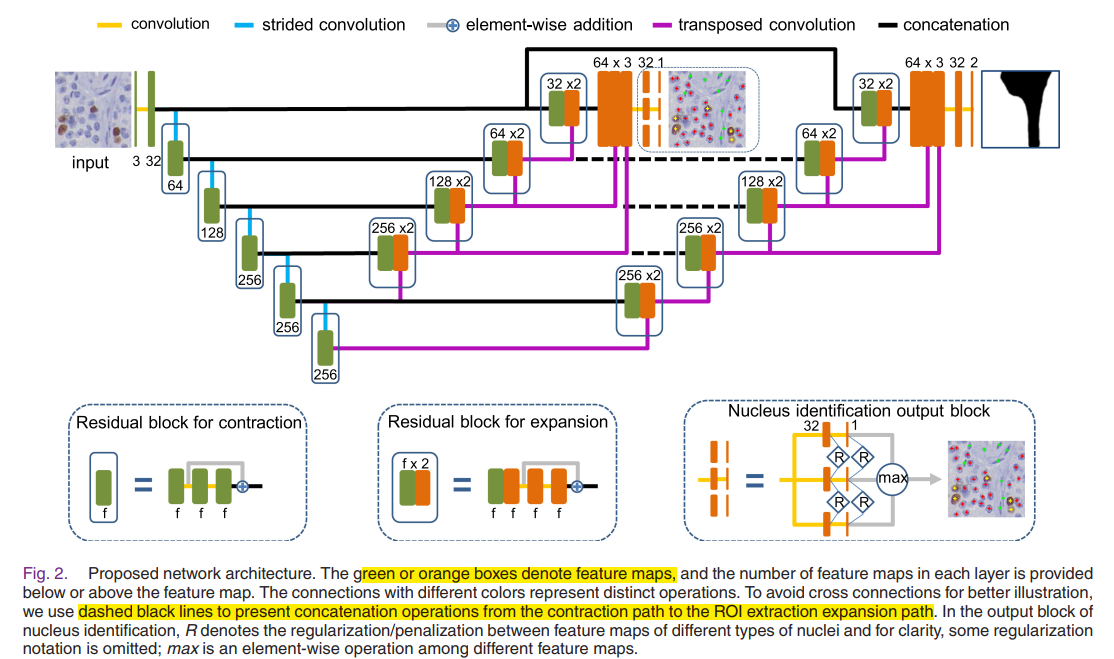
正文
组织病理学图像中的自动核识别包含单个核的检测和分类,这是非常具有挑战性的任务。 首先,由于组织切片,染色或成像特性,组织病理学图像可能具有很强的背景噪声或图像伪影。 其次,包括强度,规模和形状在内的细胞特征可能会显示出明显的类内差异,但类内差异很小,因此很难进行稳健的细胞核分类。 。 最后,原子核密集地聚集并且它们可能彼此接触甚至部分重叠是不寻常的,从而导致对单个物体进行检测和分类的边界提示不明确。
所有这些基于深度学习的方法以及上述传统机器学习方法均假定核/单元的位置是先验已知的,并且单个对象位于每个输入图像的中心,但是这种假设可能不成立。 实际应用。 在数字病理学中,单个显微镜图像通常具有成千上万个或更多的细胞核/细胞,单个对象的定位本身是一个非常具有挑战性的问题。
与以前的基于补丁的CNN方法不同,我们的模型采用任意大小的图像作为输入,并在不使用滑动窗口的情况下产生相同大小的输出。 在这种情况下,它大大降低了密集预测的计算复杂度。(这种情况下损失是如何计算的?)为了便于在最终输出图中进行对象定位,将来自收缩路径的高分辨率信息复制并连接到相应的上采样输出以进行连续学习。 请注意,展开和收缩路径可能不完全对称,因此它们的输出特征图的大小不相同,并且不适合直接按像素级联。 为了解决这个问题,我们用零填充上采样特征图,以匹配相应的下采样特征图。具有单一大小感知域的网络可能无法有效地定位具有不同比例的对象。 在组织病理学图像中,细胞核通常表现出不同的大小,以至于很难用单个感受野进行学习来捕获适当的信息以进行定位。 受[47]的启发,我们将多上下文聚合应用于来自特征图层次结构的信息融合。 具体而言,我们在不同级别的下采样特征图(对应于不同大小的接收场)上执行转置卷积,并将这些转置卷积输出连接到最后一个上采样特征图。 然后,此级联映射被馈送到输出块,该输出块由并行的两卷积(3×3)层组成,用于类特定的像素级预测,其大小与输入图像相同,并且每个对应于一个核类别 。由于此同级输出分支仅专注于ROI提取,因此它应该具有与核分类分支不同的较高层语义,尽管可以共享学习通用特征表示的较低层。FCN网络将任意大小的原始图像作为输入,并直接输出相等大小的蒙版预测。
词汇
- cumbersome 麻烦的
- pancreatic 胰
- pancreatic neuroendocrine tumors 胰腺神经内分泌肿瘤
- obviate 消除
- unary 一元
- Each curve is generated by varying ξ from 0 to 1 with an interval of 0.05
Unsupervisedly Training GANs for Segmenting Digital Pathology with Automatically Generated Annotations (2019, MIDL,年轻会议,质量较好)在无监督的情况下训练GAN以自动生成注释来分割数字病理
摘要
最近,生成对抗网络在半监督图像分析场景中展现出强大的性能。本文,作者通过为带目标形状先验知识的分割应用提出一种全监督方法。作者提出并研究了使用不同策略来生成仿真标签数据,并且使用对抗模型实现图像到图像的转换和标签领域。从实验评估看,考虑肾小球的分割,肾脏病理学的一个应用场景。实验证明提供了的概念,并且确认建立仿真标签数据的策略与稳定GAN训练有特殊的相关性。(用GAN来自动为肾小球病理图像进行分割和标注)
正文
该架构允许基于一组图像和一组(非对应)标注进行训练,只要标注是实际的(即分布与实际标注的基础分布匹配)即可。所提出的方法依赖于现实注释图像的自动生成,然后训练图像到图像的转换模型,该模型最终能够将原始图像转换为标注。 该过程基于以下假设:(1)我们需要了解标注数据的基本分布,并且需要能够对该分布进行建模(有关详细信息,请参见第2.1节)。 (2)未配对的图像到图像翻译方法需要有效地应用于图像和标注域之间的翻译。 如果直接翻译无效,则可以将其他信息添加到注释域以增强翻译过程。
令X为参考原始图像的域,令Y为标签域。 循环标准要求生成器G可以将标注蒙版转换为图像。但是,生成器F隐藏了所有低级图像细节,例如原子核和肾小管(图1),仅保留了高级图像 肾小球的形状。 仅基于这些形状,它将无法重建例如 即使图像看起来真实,位于右侧(即相同)位置的原子核也会导致较高的循环一致性损失。 考虑到这一点,我们提出并研究了第二种设置,该设置也模拟了显示低级信息的原子核。
相应的对象级别得分(Fo,Po,Ro)。 这就是说,我们将真实的正物体(即,被检测物体的中心与真实物体之间的距离小于10个像素),错过的物体和错误的正检测物体进行区分。
词汇
- glomeruli 肾小球
What do we need to build explainable AI systems for the medical domain? (2017) 我们为什么要为医学领域建立可解释性的AI系统?
摘要
一般而言,人工智能(AI)和机器学习在许多不同的应用领域中均展示出令人印象深刻的实用性成功,如自动驾驶,语音识别,推荐系统。在视觉任务中,在规模极大的数据集上训练的深度学习方法或使用强化学习方法已经超过了人类的表现,尤其是在玩游戏方面,如Atari,或围棋博弈中。甚至在医学领域中也有非常瞩目的结果。然而,这些模型的核心是他们都被当作黑盒模型,尽管我们可以理解这些模型潜在的数学原理,他们仍然缺乏明确的陈述性知识表示。这要求系统可以做出透明、可理解、可解释的决策。我们方法的最大动机是来自法律和政策方面。新的欧洲通用数据保护准则(GDPR 和ISO/IEC 27001)在2018年5月25号生效,这将使得黑盒方法很难在商业上应用。这并不意味着对自动学习方法的禁止,更像是对所有时间内解释任何发生的事物,然而,这必须使结果可以按需可追溯。这是有益的,如对于一般理解,对于教学,对于学习,对于研究,并且在法庭上可能会有所帮助。在本文,作者在可解释AI的相对较新的领域中概述了我们的一些研究主题,重点是在医学上的应用,这是一个非常特殊的领域。这是由于医疗专业人员主要使用分布式异构与复杂数据源的事实。在本文,作者专注于三个源:图像,组学数据和文本。作者认为可解释性AI的研究通常有助于促进医疗领域AI/ML的应用,特别是有助于促进透明性和可信度。
正文
一个巨大的挑战仍然悬而未决:在应用程序域的上下文中理解数据。数据的质量和恰当的特征最为重要,之前的工作显示通常组合底层特征和高层特征可以获得最佳的性能。但是,所有AI / ML成功的有效性都受到算法无法向人类专家解释其结果的限制,但这恰恰是医学领域的一个大问题。在可解释AI的上下文中,术语“理解”通常是指对模型的功能理解,与对模型的底层算法理解相反,即试图表征模型的黑匣子行为,而不必试图阐明模型的黑匣子行为。 内部运作或其内部表示形式。
两种类型的可解释性/可解释性,可以用法律中使用的拉丁名称来表示(Fellmeth和Horwitz,2009年):事后解释性=“此之后的(纬度)”,发生在相关事件之后; 例如,根据易于解释的内容来解释模型所预测的内容; 事前解释性=“在此之前(经纬度)”,发生在相关事件之前; 例如,将可解释性直接合并到AI模型的结构中,而可解释性是通过设计实现的。
在自然语言处理领域,自1970年代鼎盛以来,固有地具有更多可解释性的符号方法从未消失,但大部分已被统计,概率和最近的深度神经网络模型所取代。
词汇
- human performance 人类表现
- an explicit declarative knowledge representation 明确的陈述性知识表示
- entering into force on 对…生效
- underpin 支撑
- reenact 重新制定
- overruled 否决
This looks like that: deep learning for interpretable image recognition(2019,NeruIPS)看起来像是:深度学习以解释可识别的图像
摘要
当我们面对图像分类任务挑战时,我们通常通过解剖图像来解释原因,并且指出一个或其他类别的原型方面。每个类别的越来越多证据有助于我们做出最终的决定。本文介绍了一个神经网络结构–原型部分网络(ProtoPNET),以类似的方式说明原因:网络通过找到原型部分解剖图像,并且从原型中组合证据来做出最终的分类。模型因此以与鸟类学家、物理学家和其他可以像人们解释如何解决图像分类任务挑战中的定性方法一样。在不需要为图像部分进行任何标签下,网络使用图像层级标签来训练。本文在CUB-200-201数据集和Standford Car数据集上证明了提出的方法。实验显示ProtoPNET可以达到与非解释下部分类似的准确率,并且当有很多ProtoPNET组合到一个大的网络中时,它能达到的准确率与一个最佳性能的深度模型相当。此外,ProtoPNET提供了其他可解释性的深度模型所没有的可解释性级别。
正文
对一个训练好的CNN进行事后可解性分析是通过匹配分类性能的解释。这类分析技术包括:激活函数最大化,去卷积,显著可视化。但是这些事后可视化方法都没有解释一个模型实际上做出决策的推理过程的原因。相反,本文提出的方法建立一个基于案例推理过程,并且解释是在网络实际分类时生成的,而不是事后。
作者的工作与那些将基于注意力机制的可解释性融入CNN中。这些方法的目的在于揭露出决策时网络在输入聚焦的部分。如包括激活函数映射的注意力模型和基于不同块模型。然而,这些方法仅能指出他们注意输入中的哪一块,他们并没有向我们指出那一块与原型例子相似。另一方面,ProtoPNET不仅能揭露输入中注意的块,还能为我们指出这些块中哪一个与原型例子相似。
最近有一些研究者尝试通过衡量高度激活的图像区域和标注视觉概念之间的重叠来量化CNN中视觉表示的可解释性。然而,以数量衡量网络中的一个卷积单元的可解释性需要为特定目的网络的大规模数据集进行细粒度标注。因此,本文没有聚焦在量化网络的神经单元的可解释性,而是关注网络与人类定量相似的推理过程。
本文通过使用包含一个原型层的通用卷积来计算均方L2距离,替代卷积内积。此外,对于相同潜在训练块,约束每一个卷积滤波都相同。这个额外的约束允许我们将卷积滤波解释成可视化原型图像块,这也是重要的新颖训练过程。作者提出的网络结构如图所示
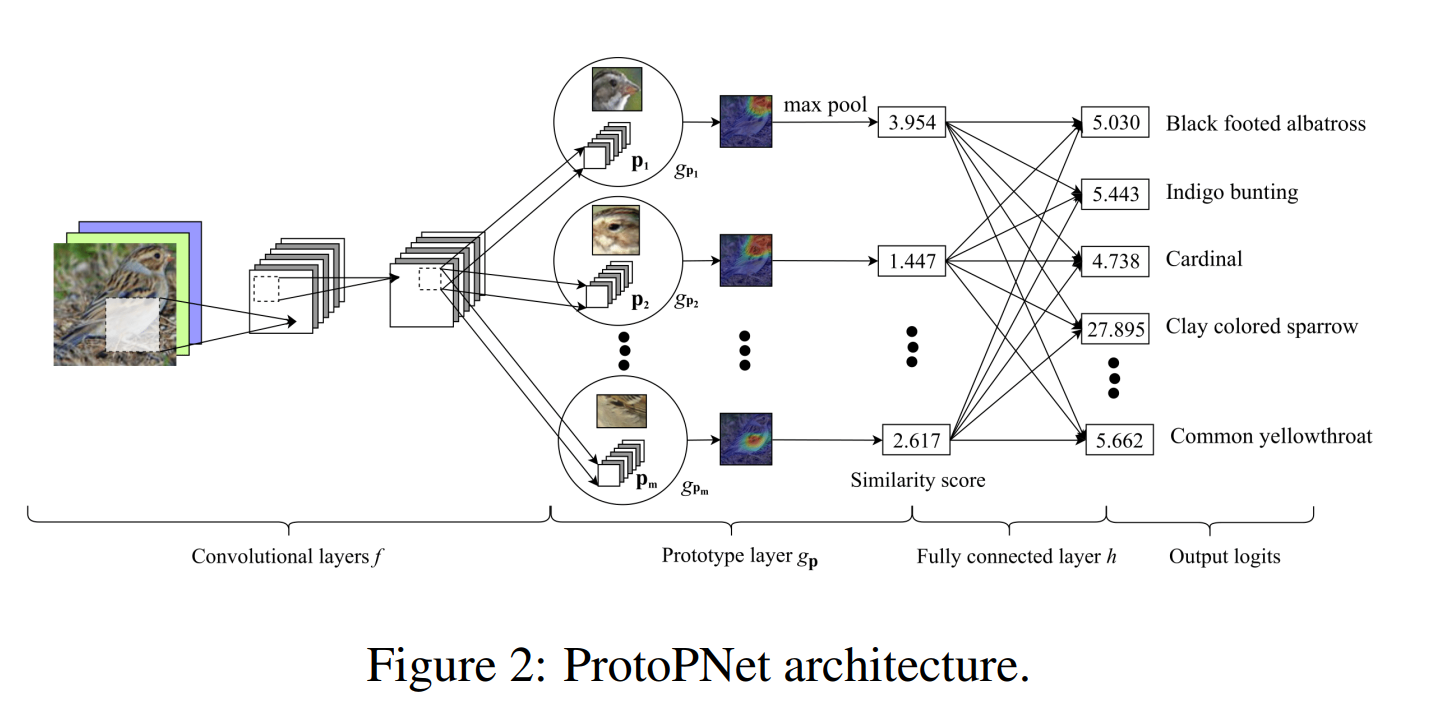
具体的推理例子如下图所示:

从直觉上说,k类原型与k类logit之间的正连接意味着与k类原型的相似性应增加图像属于k类的预测概率,而非k类原型与该类之间的负连接则增加了图像的类别。 k logit意味着与非k类原型的相似性将降低k类的预测概率。通过以这种方式固定最后一层h,我们可以强制网络学习有意义的潜在空间,因为如果k类图像的潜在斑块与非k类原型的距离太近,则会降低预测的概率。图像属于k类,并增加了训练目标中的交叉熵损失。请注意,分离成本和非k类原型与k类logit之间的负相关关系都鼓励k类原型表示语义概念,这些语义概念是k类而非其他类的特征:如果k类原型表示一个k类。语义概念也存在于对于非k类图像,此非k类图像将高度激活该k类原型,并且将因分离成本增加(即负数较少)和交叉熵增加(由于负连接而受到不利影响)而受到惩罚。分离成本对本文而言是新的,并且以前的原型学习工作尚未探讨过
结论
在这项工作中,我们定义了image处理中的一种可解释性形式(看起来像这样),该形式与人类在分类中描述自己的推理的方式一致。 我们介绍了ProtoPNet –一种适应这种形式的可解释性的网络体系结构,描述了我们的专门训练算法,并将我们的技术应用于鸟类和汽车模型识别。
词汇
- dissect 解剖
- The mounting evidence for 越来越多证据表明
- accommodates 容纳
- posthoc 事后
CELNet: Evidence Localization for Pathology Images using Weakly Supervised Learning (2019)CELNet:使用弱监督学习对病理图像进行证据定位
摘要
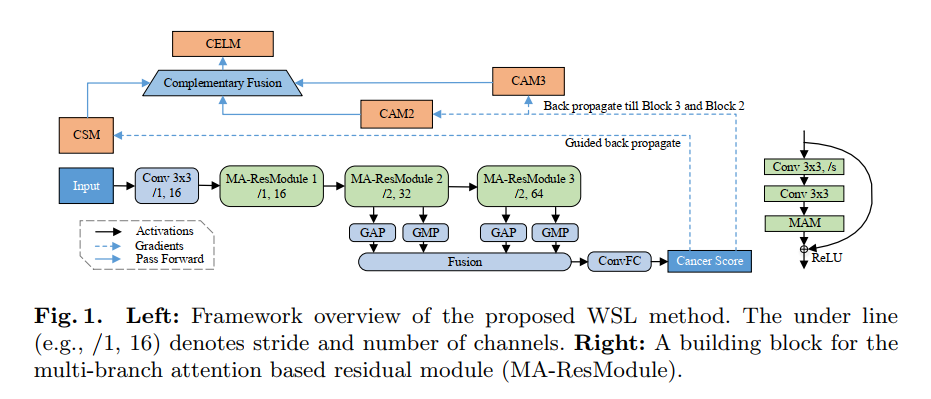
尽管深度卷积神经网络促进了数字病理学分析中图像分类和分割的性能,但它们通常在临床应用的可解释性很微弱,或需要大量的标注来取得目标的位置。为克服这些问题,作者提出了一种弱监督学习方法来从弱标注训练数据中有效地学习定位诊断标签的区分性证据。实验结果表明,我们提出的方法可以可靠地确定支持感兴趣的决策的癌性证据的位置,同时在瞥见水平和滑动水平的组织病理学癌症检测任务上仍具有竞争优势。
作者提出的网络结构如图所示
Neural Image Compression for Gigapixel Histopathology Image Analysis(2019,SCI1区(顶刊),IEEE TRANSACTIONS ON PATTERN ANALYSIS AND MACHINE INTELLIGENCE)神经图像压缩用于千兆像素组织病理学图像分析
摘要
作者提出神经图像压缩(NIC),一种两个步骤来建立卷积神经网络的方法,仅使用弱图像级别标签来进行千兆图像分析。首先,使用一个以无监督方式训练好的神经网络对千兆图像进行压缩,保留高层信息的同时抑制像素级别噪声。其次,在这些压缩图像表示上训练一个CNN网络来预测图像级别标签,避免细粒度手工标注。作者比较了几种编码策略,即重建损失最小化,对比训练和对抗特征训练,并且在一个合成任务以及两个公开组织病理学数据集上进行评估。作者发现NIC可以很好地利用图像级别标签的视觉线索,整合全局与局部视觉信息。进一步,我们可视化输入千兆像素图像中CNN感兴趣的区域,确认了这些区域与人类专家标注的区域一致。
作者提出的方法能够将相关的信息蒸馏至紧凑的图像表示。CNN能够使用这些可替代的表示进行训练,这为其他方法开辟了一条路径:大像素图像不再被认为是底层像素向量,能够以高层抽象的方式进行处理。作者在该工作用显示使用神经网络能够在潜在空间中实现分类,回归和可视化。大像素在潜在空间的表示为其他方法提供了一种思路:数据增强、生成模型、内容获取、异常检测和图像标注。
背景 具有弱图像级别标签的千兆像素图像分析的根本挑战在于这些图像中存在的低信噪比。信号通常包含与像素层级标签有关的高层和底层模式的组合,然而大多数像素都对训练无效,并且会影响模型结果。进一步的,信号的自然、空域分布是未知的,通常涉及到什么(what)以及在哪里(where)的问题。最广泛的简单假设是信号在抽象层级较低时是可识别的,如,图像层级标签有一个图像块级别的表示。通过将千兆像素图像分解成能够独立标记的图像块的方式这种方式简单的解决了what问题。但是,这种假设在不知道图像块层级表示时,是不适用的。此外,在千兆像素图像中,图像块层级的标注是一个费力,耗时长且很容易出错的过程,限制了机器学习能从人类知识学到信息的能力。其他研究假设信号能在低层的抽象存在,但不全是可识别的,如图像层级标签有一种人类标记者不了解的块层级表示。此外,在图像层级仅有这些图像块就足以进行预测,忽略图像块之间的空域排列,从而解决了where的问题。这些方法仅考虑了单独的图像块中的模式,忽略了它们之间潜在的联系。
作者提出的网络结构具体如下图所示:
总体结构
![]()
部分结构
![]()
![]()
作者没有进行与图像层级标签的视觉线索的有关的自然或空域分布的假设。作者认为CNN可以用于同时解决where和what问题。由于千兆像素计算起来是不现实的,因此作者提出一个神经网络压缩(NIC),从低层像素空间映射到高层像素空间。用这种方式,作者能够将图像压缩到一个 紧凑的表示,这使得能够用单个GPU训练CNN,以预测任何图像层级的标签。方法的关键在于:第一将千兆像素的图像分割成高分辨率的图像块;第二,每个高分辨率的图像块都使用神经网络进行压缩至低维的嵌入向量。最终,每个向量都被放入一个数组来保持原始的空域排列,这样嵌入与嵌入特征之间就保持了原始的空域信息。作者在两个数据集上分布进行了两者实验:
- Camelyon16 上进行了肿瘤转移存在性预测
- TUPAC16 上进行了基于基因表达的肿瘤增殖速度预测
使用CNN的好处在于,使用基于权重梯度的分类图能够很好的可视化CNN对输入图像感兴趣的区域。显著性图用于解释where问题。识别CNN预测的视觉证据是医学领域中很要的工作,被用于解释算法和知识发现。
很多使用公开的千兆像素图像数据集和它们使用的图像层级标签都仅包含几百个数据集点,这很容易导致过拟合。作者使用对紧凑图像进行空域裁剪,并随机取中间像素的方法训练模型。在测试阶段,旋转与紧凑图像空域维度一致的均价分布,并且对CNN的预测结果进行取平均操作。作者在WSIs图像上使用该方法。因为WSI方法中有很多区域是没有细胞的,作者检测细胞区域,采样裁剪与背景的距离成比例,以加速训练,这样就通常能采样到高细胞密度的区域用于训练。第二个方法是使用图像层面的采样增强。最终使用了能够减少参数的卷积层。
图像层级标签的可视化线索 作者使用了梯度权重分类激活图(Grad-CAM)用于训练好的CNN。该算法将对紧凑图像执行前向输入以产生3维中间表示特征向量集,其中表示第j层和第k个特征图。对于一个固定的卷积层,计算该特征图对标签y的梯度。并且对空域维度的梯度进行平均并且获得了梯度因子集,该集合表示了每个特征图对最终输出标签y的相关度。最终,将特征图与梯度因子集求积并且求和。
实验中所有的WSIs都使用了细胞分割算法来排除没有包含细胞区域。在病理图像分析中的一个常识是使用基于颜色的特征能够提供很多信息。因此,作者对输入RGB图像块的空域进行了像素密度平均以引入额外的信息以提取颜色特征。
结论
本文提出用于千兆像素神经图像压缩方法可以将相关信息蒸馏至紧凑的图像表示中。使用这些替代的学习到的表示为其他方法提供了一个新的思路:千兆图像不再被当作低级别像素数组,可以在更高级别的抽象中进行处理。在本文,作者展示了在由网络学习到的潜在层空间中分类、回归、和可视化的例子。这些可喜的结果使得在潜在空间使用更加高级的千兆享受应用成为可能,如图像增强,生成模型,内容抽取,异常检测和图像描述。
词汇
- tedious 乏味的,费神的
- excel at / good at 擅长于
- emulate 仿真
- tilted 倾斜的
- unless stated otherwise 除非另有说明
Deep Learning Under the Microscope: Improving the Interpretability of Medical Imaging Neural Networks (2019)显微镜下的深度学习:提高医学成像神经网络的可解释性
摘要
本文提出了一种新颖可解释性方法来处理组织组织病理学WSI。受功能包模型启发的深层神经网络(DNN)配备了多实例学习(MIL)分支,并针对WSI分类的弱监督训练。在不需要指导它注意的情况下,MIL避免了标签模糊并且增强了模型的表达能力。作者利用一个模型激活函数的细粒度逻辑热力图来解释决策过程。提出方法在两个组织病理学挑战数据集中进行了定量与定性分析,性能优于不同的基准模型。此外,就本文方法提供的可解释性咨询了两名病理学家,并确认了其可以集成到多种临床应用的潜力。
正文
MIL:在多样例学习(MIL)中,共享相同标签的样本被分到一个集合,视为包。将模型结构当作MIL分支合并到BagNet中。大规模病理学图像被裁剪到块来构建bag。MIL很适合这个任务,因为调整原始图像以匹配模型的输入将会导致严重的分辨率失真。因为仅对每个裁剪过的块进行图像级别的标注,将这些裁剪块组织成一个Bag,并赋予单个变迁可以避免标签的模棱两可。MIL分支对属于相同bag向量的特征维度进行平均池化,并生成向量表示整个bag。只使用bag-level标签训练MILBagnet可能导致替代稀疏。因此,最终提出的结构将会配合Single Instance Learning(SIL)和MIL分支。在一个bag前向传播到网络中后,得到SIL特征向量和MIL包级别特征向量,使用FC层对这两个特征进行处理来推断出MIL和SIL的标签。
作者提出的网络框架如图所示:
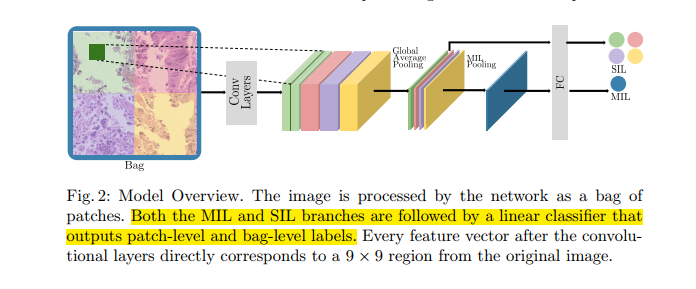
图像局部特征为主的决策:在如WSI细粒度分类任务中,图像的全局感知可能导致依赖于整幅图像中少量核的类别标签发生混淆。作者利用基于DNN的MIL来近似类别Bag-of-freatures。在模型中结合局部特征和MIL特征以规避弱监督标注引起的模糊。由线性分类器独立处理的特征允许决策过程是可追溯至每个单独的图块,以此增加可解释性。
DNN注意力的细粒度像素级别可视化:提出的结构提供了高度可解释的热力图。
提出的模型将经典BoF方法拓展至DNN,其基础模型灵感是ResNet50,模型中大量的3X3模型替换成1x1模型。这允许卷积块之后的每个2048维特征向量直接对应于输入的9x9区域。
逻辑热力图:MIL分支关闭,换成滑动窗口方法,9x9的窗口大小对图像的每个单像素处理。计算窗口的logit值,并放在每个类热力图的相应像素位置。因为Logit值衍生自图像的小区域,最高激活函数值的像素代表了模型的注意。观察注意力图,人们可以很好的理解模型的决策过程。
词汇
- false premise 错误的决策过程
- a plethora of 过多的
Pathologist-level interpretable whole-slide cancer diagnosis with deep learning (2019,nature machine intelligence)病理学家级别的深度学习可解性全切片癌症诊断
摘要
诊断病理学是识别癌症的基础和金标准。然而,在日常病理学中高观察者间的差异在实质上影响了诊断,特别是在无处不在的有效诊断医疗中心。尽管随着计算机辅助诊断(CAD)的发展,全切片病理学诊断的应用仍然不实用。然而,我们提出了一种基于人工智能的新颖病理学全切片诊断方法,来解决诊断中缺乏的可解释性。提出方法可以模仿人类诊断推理过程并将千兆像素直接转换成一系列可解释性预测,提供了第二种意见并且鼓励在临床种达成共识。此外,使用913个收集样本的膀胱癌病人的全切片数据集。实验结果显示,在尿路上皮癌的诊断性能可以媲美17个病理学专家。作者详细提出方法提供了一个新的创新并且对于诊断建议很有意义,也能够以低代价部署到下一个迭代产品种,AI加持的CAD技术用于诊断病理学。
作者提出的方法包括三个神经网络:
- 扫描网络(s-net):用于检测肿瘤区域;自动选择在被检测到肿瘤附件的组织图像集合作为诊断的ROIs区域。
- 诊断者网络(d-net):当描述观察时,对每个ROI提取特征,通过描述病理学特征(显微镜发现)来分析解释每个ROI,并且展示了网络注意的特征来解释网络学到的东西。所有ROIs的信息编码至低维特征表示集合。
- 聚合器网络(a-net):聚合所有特征并且做出最终诊断。
网络结构如下图所示:
作者使用图像描述语来解释神经网络的决策过程(LSTM实现)。
词汇
- carcinomas 癌
Multimodal Multitask Representation Learning for Pathology Biobank Metadata Prediction(2019)病理生物库元数据预测的多模式多任务表示学习
摘要
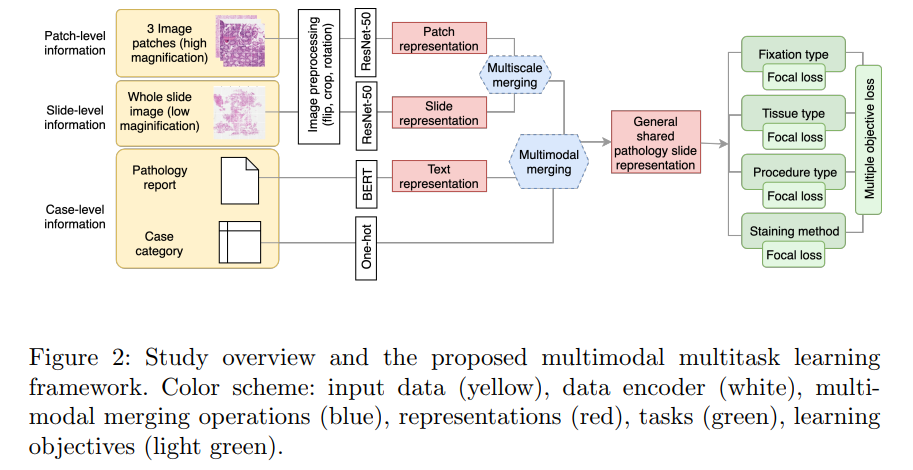
元数据是经过精心整理和紧凑格式的一般特征数据,并且已被证明可用于决策制定,知识发现以及生物库的异构数据组织。在生物库的所有数据类型中,病理学是生物库中的关键组件,并且作为诊断的金标准。为最大化生物库的利用,促进生物医疗科学的快速进步,必须使用填充良好的病理学元数据来组织数据。然而,这些信息的手工标记是繁琐且耗时的。本文提出了一个多模态多任务学习框架来预测四种病理学图像中的四个主要切片级别元数据。提出的框架学习跨组织切片、病理学报告和用例级别结构化数据的通用表示。与单模型单任务基准相比,作者证明了使用提出的方法在两个测试集(一个数据集来自同源的TCGA另一个是留一测试集(TTH))中的四个任务上均能获得性能上的提升。在测试集中,在两个数据集中的四个任务平均ROC曲线的性能提升为16.48%和9.05%。通过更好地利用病理学生物库,该病理学元数据预测系统可能可以用于减轻专家标注的工作并且最终加速数据驱动的研究。
网络结构如下图所示:
正文
在生物医学领域,元数据是数据封存和其他下游数据驱动应用的关键。其中著名应用之一就是生物库的发展。生物库是一个存储用于研究使用的生物组织样本的资料库,开发一个生物库的实质是利用人口稠密的元数据来组织一个有组织的数据集。生物库在很多科学和临床突破种扮演者很重要的角色。生物库通常包含从不同异质格式形式的大量数据,如基于序列、病理学图像和临床报告。为了使用这些数据集,拥有人口稠密的元数据是必须的。
在生物库中,病理学元数据与长期随访生存记录对于建模疾病处理与病人结果预测非常有价值。然而,它们通常也是组织最少的数据。这主要是因为机构间数据封存过程的差异以及现代病理学中生物组织的人工标记。
本文研究了多模态多任务学习用以联合预测同时共享的图像、文本、和有限结构化类别变量以及样本尺寸模式不平衡的多切片元数据。作者合并了病理学报告的样例级别与切片级别组织图像,因为这些存储了链接到不同元数据的不同信息。本文的贡献如下:
- 提出了一种使用图像、自由文本与结构化数据的多模态多任务学习框架,该框架用于有限不平衡的病理学元数据预测。
- 在四种病理学元数据预测任务中,提出的多模态多任务框架优于基准单模态单任务框架。
- 观察到通过在多任务框架顶层添加多模型信息的协同效果优于仅使用多模态或多任务。
多任务学习MTL:作者认为多任务学习通过训练模型来从共享表示中同时预测相关的病理学元数据,从而有助于利用来自其他任务中获得监督信号。该设计引起的自然问题就是如何进行参数共享呢?MTL学习共享表示的主要策略有两个,硬参数共享和软参数共享。
- 硬参数共享策略:只有一条从输入到共享表示的单路径,后跟与任务相关的独立参数的表示。这允许底层参数与每个特定任务相关参数进行共享。这允许学习一个通用的共享表示同时最优化下游任务。
- 软参数共享策略:每个任务有其从输入到输出的路径。通过施加一个联合正则化实现不同路径的软共享。这允许不同表示在不同任务路径之间具有一定程度的相似性,而不必强制它们相同。
除参数共享外,MTL中其他关键是跨不同任务的损失权重。本文合并硬参数共享策略与基于梯度的多目标优化来学习更好跨任务共享的表示。
多模态学习:多模型学习的目标是学习一种数据表示,该数据表示可以从跨不同模式的数据中捕获共享和独立的信息。这种方法非常适合用于在生物医疗领域,这是因为病人数据通常有多种模型组成,如波形、索赔数据、自由文本、图像和基因序列。研究者利用来自不同模态的信息来解决不同生物医疗问题,如模式识别、报告生成、医学语言翻译,临床事件预测。然而,多模态学习的关键挑战在于跨不同模型的异质性,这是由于大量不同统计属性和不同级别的噪声。本文中,作者探索不同策略来有效合并异质性模态。多模态学习有两种主要的方法:共享表示学习和跨模态协同表示学习。
- 共享表示学习:如 学习通用的嵌入空间,迫使模型从多模态数据中学到单一潜层表示。
- 跨模态协同表示学习:或嵌入对齐,使用一个额外的步骤来对齐表示的几何结构相似性假设下的不同模态的表示。
共享表示学习,早期融合和后期融合是两种最突出的方法。
- 早期融合(early fusion):有助于捕获模态间底层表示,当不同模型中的要素之间存在依赖关系时,这是理想的选择。
- 后期融合(late fusion):构建一个元分类器来保留更多的单模态信息,因为这种方法不会在低级别上对模态之间的交互进行建模。
作者的目的是从异质病理学图像、文本和结构化数据中学习通用表示,使用底早期融合策略来捕获模态间的底层交互并且尽可能保持其泛化性。研究了使用不同操作实现模态融合的模型性能差异。
使用ResNet和BERT分别提取图像和文本特征。对于如原发癌部位,用例级别结构化数据被编码成one-hot格式。多级别数据的目的在于将先验知识融合到模型用以减少预测搜索空间。例如,使用原发癌位置作为输入帮助模型聚焦到更少的组织类型,可能出现无需从病理学层面数据泄露问题的样例中。
数据预处理:所有的千兆像素病理学图像使用0.312*image,然后不管长宽比,使用双线性插值rescale到512x512。
在这四个预测任务中,组织类型是病理学家最具挑战性的任务。 为了从图像中对组织类型进行分类,病理学家将需要在低倍率和高倍率下检查组织形态,同时结合病理报告中有关病例的先验知识。观察到性能的主要增益来自于合并多模态而不是多任务。我们认为性能较差是由于补丁图像与文本/结构化数据之间的信息相似性所致。 主要由于图像噪声和补丁采样噪声,补丁图像预计会比其他两种方式更嘈杂。 补丁图像仅包含狭窄的特定区域。 因此,噪声可能来自捕获具有在不同组织类型中常见的相似视觉特征的组织图像。
词汇
- curate 受托人
- The Cancer Genome Atlas (TCGA)
- synergistic 协同作用
- coordinated representation 协同表示
- multitask learning( MTL) 多任务学习,联合学习,附属任务学习
- head with 与
- aspect ratio 长宽比
Computer aided quantification of intratumoral stroma yields an independent prognosticator in rectal cancer (2019,SCI二区)计算机辅助量化肿瘤内基质可产生直肠癌的独立预后因子
摘要
目的:肿瘤基质比例(TSR)在大肠癌和其他固体癌症中是一个独立的预后因子。最近日常组织诊断中数字病理学的引入为自动TSR分析带来新的机遇。作者调查了在直肠癌全切片图像中肿瘤基质的计算机辅助量化的潜在可能。
方法:两位专家对129个直肠腺癌患者的组织病理学切片进行分析,选择了合适的基质热点并通过视觉方法评估了TSR。训练了一种基于深度学习的半自动化方法,以对直肠癌组织学中的所有相关组织类型进行细分,然后将其应用于专家提供的热点。通过两种TSR方法(视觉和自动化)将病人分成“高基质”和“低基质”两种。这可以进行特定疾病与疾病无关生存事件的预后比较。
结果:将低基质作为基准,发现自动化TSR的预后独立于年龄、性别、pT-stage,淋巴结点状态、肿瘤得分,并决定是否给予辅助治疗。特定疾病生存(hazard ratio = 2.05)和疾病无关生存(hazard ratio = 2.05)。在多变量分析中,可视化评估TSR没有显示出该特点。
结论:该项工作表明在在由用户提供的基质热点评估中,直肠癌中TSR是一种独立预后因子。此处介绍的基于深度学习的技术可能对病理学的例行诊断提供重要帮助。
词汇
- colorectal cancer 大肠癌
- rectal cancer 直肠癌
- rectal adenocarcinoma 直肠腺癌
- hazard ratio 危险几率
Development and validation of a deep learning algorithm for improving Gleason scoring of prostate cancer (2019,SCI医学1区顶刊)用于改善前列腺癌的Gleason评分的深度学习算法的开发和验证
摘要
对于前列腺癌症患者而言,Gleason评分是最重要的预后因子之一,可能独立于阶段确定治疗。然而,Gleason评分是基于肿瘤形态学的主观显微镜检查,可再现性差。作者提出了一种用于前列腺切除手术的全切片图像的Gleason评分的深度学习系统。作者的系统使用了从1226个切片得到的1.12亿张病理学标注图像块,并且在331张独立的验证集切片上进行评估。与由泌尿生殖系统病理学专家提供的参考标注相比,验证集中29位普通病理学专家的均方准确率位0.61。系统能够达到较大的性能提升到0.70,并且与临床随访数据的相关性倾向于更好的患者风险分层。本文方法可以提高Gleason评分和后续决策治疗的准确率,特别是当缺少专家时。系统性能超过了当前Gleason系统,可以更精细地表征和量化肿瘤形态,为完善Gleason系统本身提供了机会。
正文
前列腺癌症是男性中第二常见的癌症,一生中大约有九分之一的男性被诊断为癌症。几乎在所有大型前列腺癌结果研究中,格里森评分和肿瘤分期仍然是影响预后的最有力指标。尽管格里森评分在预后和患者管理方面具有无可争议的作用,但病理学家对格里森的评分是一种主观的锻炼,其观察者间和观察者内变异性均不理想,据报道格里森评分不一致的范围为30%至53%。这项研究通过应用深度学习对整个临床标本进行Gleason分级,对先前的研究进行了补充,并且还重要地使用了独立的参考标准来将算法的准确性与经董事会认证的病理学家进行比较。
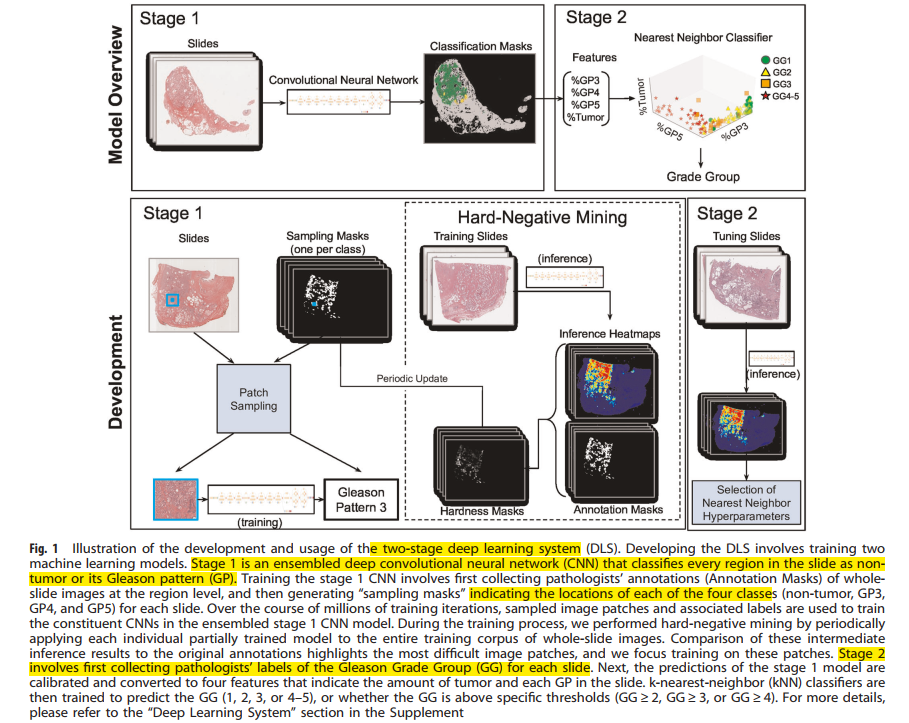
本文提出的深度学习系统包括两个阶段
- 阶段一是集成的深度CNN网络对切片中的每个区域分类成非肿瘤或者Gleason模式(GP)。训练CNN首先要收集病理学家在区域级别对全切片图像的标注(标注掩膜),然后生成“sampling masking”来表示每张切片中四种类别(非肿瘤,GP3,GP4,GP5)的位置,经过数百万次训练迭代,采样好的图像块与其对应的标签被用于训练构建好的集成的阶段一的CNN模型。在训练过程中,通过定期将每个单独的部分训练的模型应用于整个全切片图像的整个训练语料库来执行硬负挖掘。将这些中间推断结果与原始标注相比,找出最困难的图像块,然后集中训练这些块。
- 阶段二涉及首先收集每张切片的Gleason Grade Group的病理学标注。然后阶段1模型的预测结果被校准和转换到四种特征,以表示切片中肿瘤数量与每个GP。随后,KNN分类器被用于预测GG,或者GG是否高于特定阈值。
作者提出的网络结构如图所示
词汇
- Over the course of millions of training iterations 经过数百万次实验迭代
- censorship 审查制度
- a cohort of 一群
- concordant 一致的
- concurs 同意
- Substantial 重大的
- cusp 尖顶
- antigen 抗原
Cytokeratin-Supervised Deep Learning for Automatic Recognition of Epithelial Cells in Breast Cancers Stained for ER, PR, and Ki-67(2020, SCI1区顶刊 TMI)细胞角蛋白指导的深度学习,可自动识别ER,PR和Ki-67染色的乳腺癌中的上皮细胞
摘要
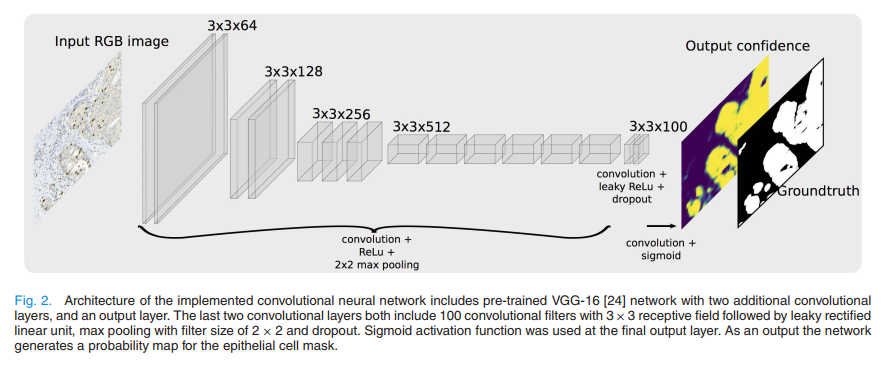
ER,PR,和Ki67的免疫组织化学(IHC)被用于日常的乳腺癌诊断中。染色细胞(标注索引)比例的确定应该限制在恶性上皮细胞,仔细避免肿瘤浸润基质与发炎细胞。本文开发了一种基于深度学习的数字掩膜,用以使用荧光色细胞角蛋白Ki67双重染色和苏木精-IHC连续染色的上皮细胞自动检测。使用由152位病人样本的侵入性乳腺癌的图像块微调预训练深度CNN网络的部分层。从98个未见过的样本中捕获的366张图像,研究了训练好的数字上皮细胞掩膜,通过比较上皮细胞蒙片和细胞角蛋白图像,以及由两名病理学家对明亮图像进行视觉评估。得到一个好的上皮组织区分性能(AUC of mean ROC=0.93),并与病例学专家的视觉评估一致。上皮细胞掩蔽对Ki-67标记指数的影响是可观的。 在应用基于深度学习的上皮细胞遮罩后,最初被分类为低增殖(Ki-67 <14%)而没有上皮细胞遮蔽的52个肿瘤图像被重新分类为高增殖(Ki-67≥14%)。数字上皮组织掩膜被发现同意适用于IHC,ER,PR。作者认为深度学习可以应用于检测传统亮度场IHC染色的乳腺癌中的癌细胞。
正文
ER、PR、Ki67的IHC染色被常用于乳腺癌诊断评估。尽管PR与ER分析饱受争议,但它们的在当前诊断的角色已经建立。肿瘤细胞增殖率,由Ki67免疫染色定义,是定义病人诊断的额外工具。正(棕色)染色核(或像素)占总核(棕色核+染色的核)的比率作为labeling index。
作者提出的网络结构如图所示
词汇
- epithelial 上皮
- inflammatory 发炎的
- cytokeratin 细胞角蛋白
- low proliferation 低扩散
MITOS-RCNN: A Novel Approach to Mitotic Figure Detection in Breast Cancer Histopathology Images using Region Based Convolutional Neural Networks (2018)MITOS-RCNN:基于区域卷积神经网络的乳腺癌组织病理学图像中有丝分裂图检测的新方法
摘要
研究表明,仅2018年一年,就有266,120例新发浸润性乳腺癌病例和40,920例乳腺癌诱发的死亡病例。尽管这种疾病无处不在,但是当前获得准确的乳腺癌预后的过程既繁琐又耗时,需要训练有素的病理学家手动检查组织病理学图像才能鉴定出表征各种癌症严重程度的特征。本文提出MITOS-RCNN:一种新颖的基于区域的卷积神经网络(RCNN),适用于小物体检测,以准确地对由诺丁汉评分系统描述的表征肿瘤特征性的三个因素之一进行分级:有丝分裂计数。 其他有丝分裂图计数和检测的计算方法不能证明召回率或精确度在临床上是可行的。 我们的模型优于最近参加ICPR 2012挑战赛,AMIDA 2013挑战赛和MITOS-ATYPIA-14挑战赛的所有参与者,以及最近发表的工作。 我们的模型的F-measure得分为0.955,与先前提出的最精确模型相比,其准确性提高了6.11%。
正文
我们将专注于准确提取乳腺癌预后的最有据可查和最显着的特征:有丝分裂计数。 有丝分裂计数几乎不需要专业解释,由于使用每个高倍视野的有丝分裂计数(HPF’s:在显微镜的最大放大倍数下可见的区域)来确定增殖率的简单指标:每10个HPF’s有0-9个有丝分裂是低增殖,每有10-19个 10 HPF是中等程度的增殖,每10 HPF超过19个有丝分裂是严重增殖。
方法(提出模型):
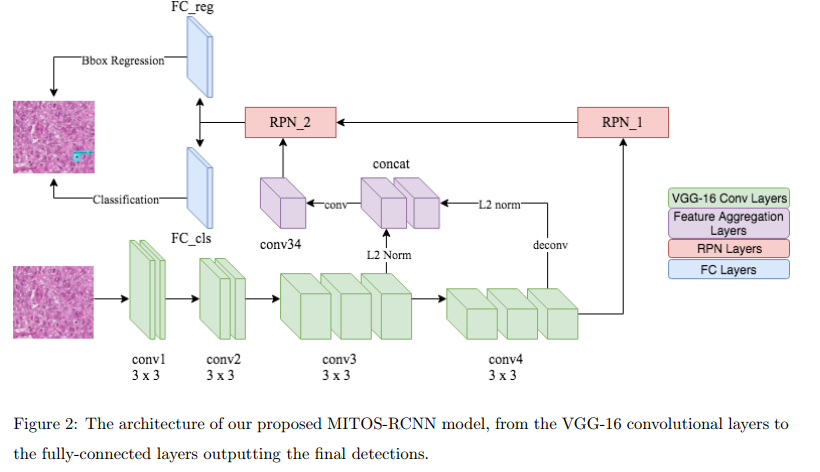
- 小物体检测:采用更小尺寸目标框的Faster-RCNN,从标准的平均44px降到30px,并且针对不同层指定最小检测目标尺寸为15px和22px(分别针对conv3和conv4)。
- 两阶段自上而下级联多层级提取生成:这可以让模型使用从高层和底层特征的语义知识来实现准确的目标检测。RPN1跟在conv4后,并生成15kproposals(极大值抑制阈值为0.7)。RPN2利用conv3和conv4拼接构成的特征图。为了实现拼接,使用上采用层对conv4特征图进行放大以匹配conv3的分辨率,然后对每层使用L2norm进行规范化,然后进行拼接,拼接特征最终被映射到256x1x1大小。RPN2的输入来自两个源:RPN1的proposals和在conv3+4特征图上使用滑动窗得到64^2px和1:1的长宽比。最终使用阈值为0.7的极大值抑制进行提纯,后喂入ROI池化层得到规范化后的proposal尺寸区域。
- 检测网络:检测网络由两个兄弟输出层构成:一个边界框分类层和一个边界框回归层。
Terabyte-scale Deep Multiple Instance Learning for Classification and Localization in Pathology (2018)TB级深度多实例学习,用于病理学中的分类和本地化
摘要
在计算病理学领域,由于缺少规模标注数据集限制了SOTA深度学习解决方法赋能决策支持系统的能力。直到最近,仅依赖于几百张切片的数据集的研究,不足以训练可以在临床上大规模使用的模型。作者收集了12160切片构成的数据集,在病理学上比以前的数据集大两个数量级,相当于整个ImageNet数据集的像素的25倍。给定数据集的大小,作者有可能在多实例学习(MIL)假设下训练深度学习模型,该模型只需要对整体切片诊断即可进行训练,避免了通常是监督学习一部分的所有昂贵的像素化标注方法。作者在复杂任务中测试了其框架,针活检前列腺癌症诊断。作者在留一测试集的1824张切片上进行了ML管道性能的全面的验证,得到AUC的0.98的结果。这些结果为大规模训练准确的诊断预测模型开辟了道路,为在临床中部署决策支持系统奠定了基础。模型的预测性能取决于数据集的规模。
正文
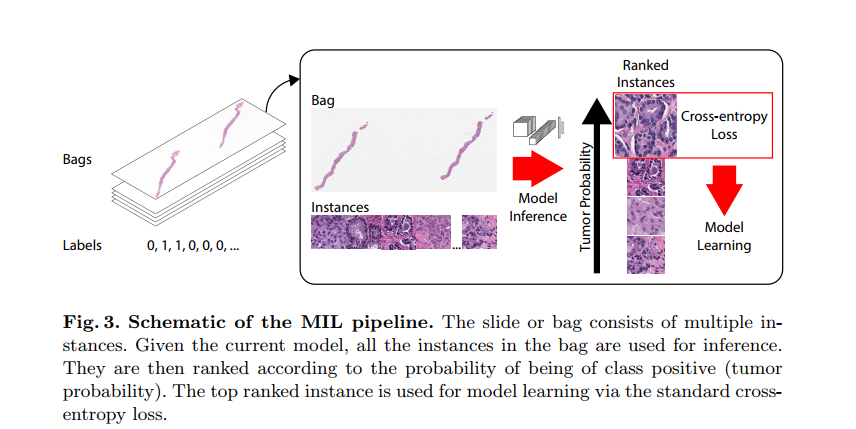
当仅知道切片级别的类别而未知切片中每个片的类别时,可以在经典的MIL范式下形式化基于图块级别分类器的整个数字幻灯片的分类。正性bag:一个bag中至少有一个例子被分类成正性。负性bag:bag中所有得实例都得被分类成负性。方法的完整过程由以下步骤构成:
- 数据集中每个切片的堆;对于每个轮次,整个训练数据集都需要过一遍。通过算法,切片中所有背景都被有效的丢弃,极大减少了每张切片的计算量,绝大部分都没有被组织覆盖。可以在不同的放大倍率下执行平铺,并且相邻图块之间的重叠程度可以不同。
- 在整个数据上进行完整的推理
- 实例切片内排名
- 基于每张切片的排名前1位的实例进行模型学习
作者提出的网络结构如下图所示
词汇
- in the order of 按照…的顺序
- on needle biopsies 针活检
- lesions 病变
- reiterate 重申
- indispensable 基本的
TOWARDS UNSUPERVISED CANCER SUBTYPING: PREDICTING PROGNOSIS USING A HISTOLOGIC VISUAL DICTIONARY(2019,MICCAI医学顶会)走向无监督癌症亚型:使用组织病理学视觉词典来预测预后
摘要
不同于通常的癌症,如前列腺癌与乳腺癌,由于样本过小,稀少癌症的肿瘤评级非常困难并且难以定义。完成这项任务所需大量时间,以及难以提取人类观察的模式。最具挑战性的例子之一是肝内胆管癌,由胆系统引起的原发性肝癌,其具有公认的肿瘤异质性,没有分级范例或预后生物标志物。本文提出一种新的无监督基于深度卷积学习自编码器的聚类模型,基于视觉相似性,模型对246张ICC数字全切片图像聚类成细胞组和肿瘤结构化形态。从该病理学视觉字典,作者使用聚类作为协变量来训练考克斯比例风险生存模型。在协变量分析中,三种聚类与自由复发生成高度相关。三种聚类的组合对于多协变量分析非常重要。在所有聚类的多协变量分析中,5个聚类显示出与协变量生存高度相关,然而所有模型没有测量是显著的。最终,一位病理学家将临床术语分配给了视觉词典中的重要词类,并发现了支持以下假设的证据:胶原蛋白丰富的纤维化在疾病严重程度中起作用。这些结果为未来癌症亚型提供了洞察力,并且显示计算病理学有助于疾病预后,特别是稀有癌症。
正文
癌症分级是预测疾病预后和治疗方向的非常重要的工具。常见癌症,如乳腺癌和前列腺癌,已经建立的评级机制在大规模样本中得到验证。前列腺癌等级系统是癌症医学的最重要的国际接受的评分方案。由唐纳德·格里森(Donald Gleason)开发的该系统称为格里森评分系统(GGS),该方法将成千上万例病例的病理学观察到的组织学模式与结果数据相关联。 经过近二十年的反复设计和验证该评分得预后分类,他于1992年发表了对前列腺癌分级的最终综述。此后,它成为临床标准[2],在2005年和2011年由国际专家学会再次进行小幅修订 。 尽管前列腺癌分层的金标准,但GGS仍需进行持续的严格评估。
为某种稀有癌症开发一种评级系统构成了一系列挑战。ICC是胆管癌症的一种,在US大约10万人中有一个人患该病,并且数量正在不断上升。目前对于该疾病,没有能被组织病理学广泛接受的亚型或评级系统。对ICC进行分型的一个主要限制因素是每个研究机构只能获得少量病例
急切需要一种来从稀有癌症有限组织学数据集中有效识别预后相关细胞和结构化形态来构建危险分层系统,目前在许多癌症类型中都缺乏。理性情况下,这些系统应该利用全面且可复现的组织学模式的视觉系统。一旦生成以后,视觉系统必须转化成能被病理学家们普遍理解的组织病理学术语。
方法
作者收集了大量的全切片数据,除去数据集中失焦图像后,训练一个而分类器。训练该分类器时,为了使正负样本数量保持相对平衡,对部分非模糊样本构建其负样本。0代表锐化(正常)样本?
本文提出的聚类算法利用ResNet18作为编码器,解码器由5个步长为1的卷积层构成,为了保证特征图的尺寸,在每个卷积层之前都是用上采样层。没有使用bn层。模型需要优化的参数有两个:网络的权重和聚类中心的位置,即嵌入空间的质心C。初始化时为每个样本指定有一个随机的聚类,质心C通过公式计算。最终的聚类中心C*通过最小化编码嵌入空间与上一次C值的差异得到。
作者提出的网络结构如下图所示:
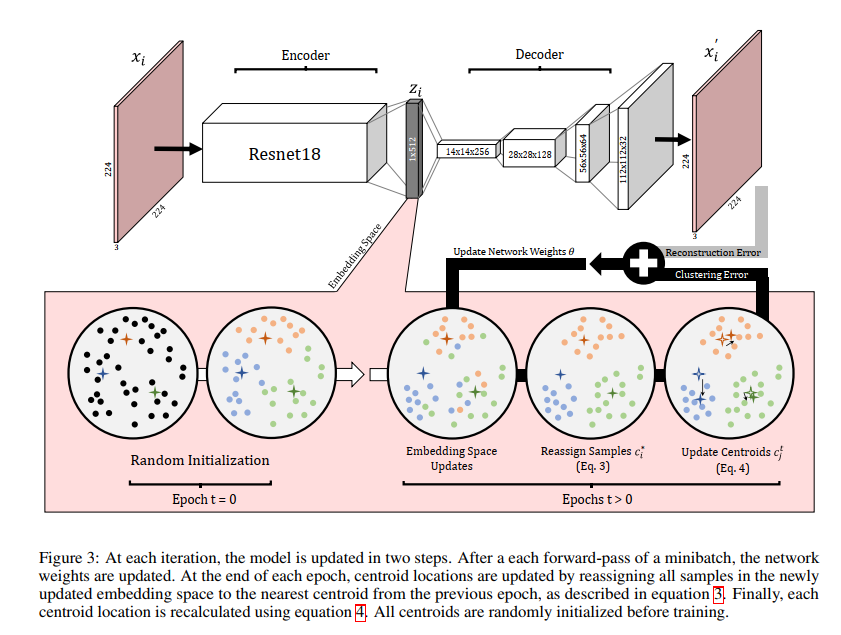
词汇
- the sheer volume of 纯粹的数量
- inherent 固有的
- intrahepatic cholangiocarcinoma ICC, 肝内胆管癌
- the biliary system 胆系统
- primary liver cancer 原发性肝癌
- well-recognized 公认的
- covariates 协变量
- Cox-proportional hazard survival models. 考克斯比例风险生存模型。
- recurrence-free 自由复发
- parenthesis 插入语
- bile duct 胆管
- thumbnail 缩图
- Collagen 胶原
