【论文阅读(三)】TransMed Transformers Advance Multi-modal Medical Image Classification
导语:本篇文章发表在IEEE ACCESS, 是第一个将transformer用于医学图像分类中的工作。作者利用了CNN和transformer的优势,建立了一个符合多模态医学图像的长期依赖关系的模型。
摘要
在过去的十年中,在医学图像分析任务中,CNN的性能显示出很强竞争力,如疾病分类,肿瘤分割,病灶检测。CNN在提取图像的局部特征时显示出很大的优势。然而,由于卷积操作的局部性,它无法很好地处理长范围关系。最近,transformers被应用到计算机视觉中,并且在大规模数据集上取得了令人瞩目地成功。与自然图像相比,多模态医学图像有清晰和重要的长期依赖。有效的多模态融合策略可以极大地促进深度模型的性能。这敦促我们研究基于transformer结构,并将其应用到多模态医学图像中。目前基于transformer的网络结构需要大规模数据集来获得更好的性能。然而,医学图像数据集相对较小,这使得该领域很难应用完全的transformer来进行医学图像分析。因此,作者提出TransMed来进行多模态医学图像分类。TransMed融合了CNN和transformer的优点,可以有效提取图像中的底层特征,并建立模态间的长期依赖。作者在腮腺肿瘤预诊断这个具有挑战性的任务中评估了作者的模型,结果证明了模型的有效性。作者认为在大规模医学图像分析任务中,CNN和transformer的组合具有巨大的潜在优势。
背景
Transformer首先被用于NLP领域。它是一种主要基于自注意机制的深度神经网络,用于提取固有特征。由于其强大的表示能力,研究者希望将找到一种将transformer应用到计算机视觉任务的方式。与文本相比,图像涉及大尺寸,噪声和冗余模态,因此在图像任务中使用transformer将会更困难。最近,tranformer在计算机视觉中取得了重大突破。提出了大量基于transformer的计算机视觉方法,如目标检测的DETR,语义分割的SETR,图像识别的ViT和DeiT。
transformer在自然图像中取得了成功,但是在医学图像分析中受到的关注却很少。多模态广泛的存在于医学图像分析,以实现病灶分割和疾病分类。目前基于深度学习的医学图像多模态融合可以分类成三种:输入层级融合,特征层级融合,和决策层级融合。
- input-level fusion: 由多通道的方式将多模态图像融合到深度网络中,学习融合特征表示,然后训练网络。该策略可以最大限度地保留原始图像信息并且学习图像特征。但是,该策略很难建立起同一个患者的不同模态之间的内在联系,这将会降低模型的性能。
- feature-level fusion: 将图像地每个模态单独作为输入,训练单个深度网络。每个表示在网络层融合,最终融合结果输入决策层来获得最终结果。特征融合网络可以有效地捕获同一病人的不同模态信息。但是,特征层级网络的每个模态都有一个相应得网络结构,这会带来巨大的计算代价,特别是在模态数据很多的情况下。
- decision-level fusion: 整合每个网络的输出来获得最终的结果。该策略目的在于从不同的独立的模态中学习更加丰富的信息。但是,决策层级融合的每个模态输出都彼此独立,因此模型无法建立相同患者不同模态之间的内在联系。同样计算复杂度很高。
因此,有效组合这三种融合策略非常迫切。一个好的多模态融合策略应该尽可能以低的计算复杂度实现不同模态之间的交互。与CNN相比,transformer可以有效地维持序列之间的长期依赖。目前的基于transformer的计算机视觉模型主要处理2D自然图像,如ImageNet和其他大规模数据。在2D图像中构建序列的方法是将图像分割成一系列的图像块。这种类型的序列构建方法隐式的现实了长期依赖,但不是非常直接地,因此可能很难带来重大地性能提升。
与此相反,在医学图像中存在更加显性的序列,因为它包含重要的长期依赖和语义信息。如图所示:
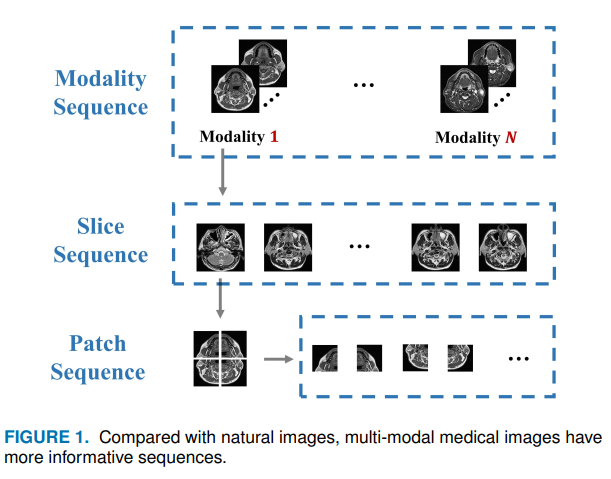
由于人类器官的相似性,大多数视觉表示在医学图像中是有序的。销毁这些序列将会严重地减少模型的性能。与自然图像相比,医学图像(模态,slice, patch)的序列关系保留更多的丰富信息。实际上,医生也会合成每个模态的病理学信息来做出诊断。然而,当前的多模态融合方法过于简单地考虑这些序列之间的联系,并且缺乏对长期依赖的建模。transformer是一种处理序列关系的优雅,有效地并且强大地编码器,这激励着作者提出基于transformers的多模态医学图像分类方法。
在本文作者提出了第一个研究来探究医学图像分类背景下transformer的巨大潜力。受到transformer在提取序列之间关系非常有效。然而,由于医学图像数据集规模较小以及缺乏足够的信息来创建底层语义特征之间的关系,在多模态医学图像分类中,基于ViT和DeiT的纯transformer网络的的性能并不理想。因此,作者提出了TransMeD, 组合了CNN和transformer的优点来捕获底层特征和跨模态高层连接。TransMed首先将处理多模态图像处理成序列并将他们送入CNN中,然后使用transformer来学习序列之间的联系并做出预测。由于transformer可以有效建模多模态图像的全局特征,TransMed在性能、运行速度和准确率上均优于当前的多模态融合方法。大量实验证明了模型的有效性。
总而言之,作者指出了3个贡献:
- 首次将transformer应用于医学图像分类,并且大大提升了深度模型的准确性和效率
- 提出了一种新颖的多模态图像融合策略,该策略可以有效捕获图像的不同模态之间的互信息
- 实验验证证明了提出方法可以获得腮腺肿瘤分类的性能提升。
相关工作
多模态医学图像分析
多模态医学图像分析是医学图像分析中基本和最具挑战性的任务。不同模态合理的融合可以潜在地增强深度网络。多模态融合可以捕获更丰富地病理学信息和提升诊断地质量。在多模态医学图像分析中,输入层级融合是最常见地融合方法。
TRANSFORMERS
transformer首先被提出用于机器翻译并且在大量地NLP任务中取得了令人满意地结果。然后,transformer被应用到计算机视觉领域,并且进行了一些改进。结果现实了其性能可能会超越纯CNN的性能。一些工作同时使用CNN和transformer的结构,一些工作直接使用纯transformer替代CNN。在计算机视觉领域,这些方法显示出令人鼓舞的结果,但是这些方法直接用于多模态医疗图像效果不佳,且需要大量的计算资源。
方法
多模态医疗图像分类是最直接方法在于直接训练CNN(例如ResNet)。首先,图像被编码成高层特征表示,并且这些特征和决策被融合做出决策。不同于现存的方法,作者使用transformer将自注意机制介绍入多模态融合策略。作者将介绍如何直接将transformer应用到分解的图像块中获得聚合特征表示。整体框架入下图所示:
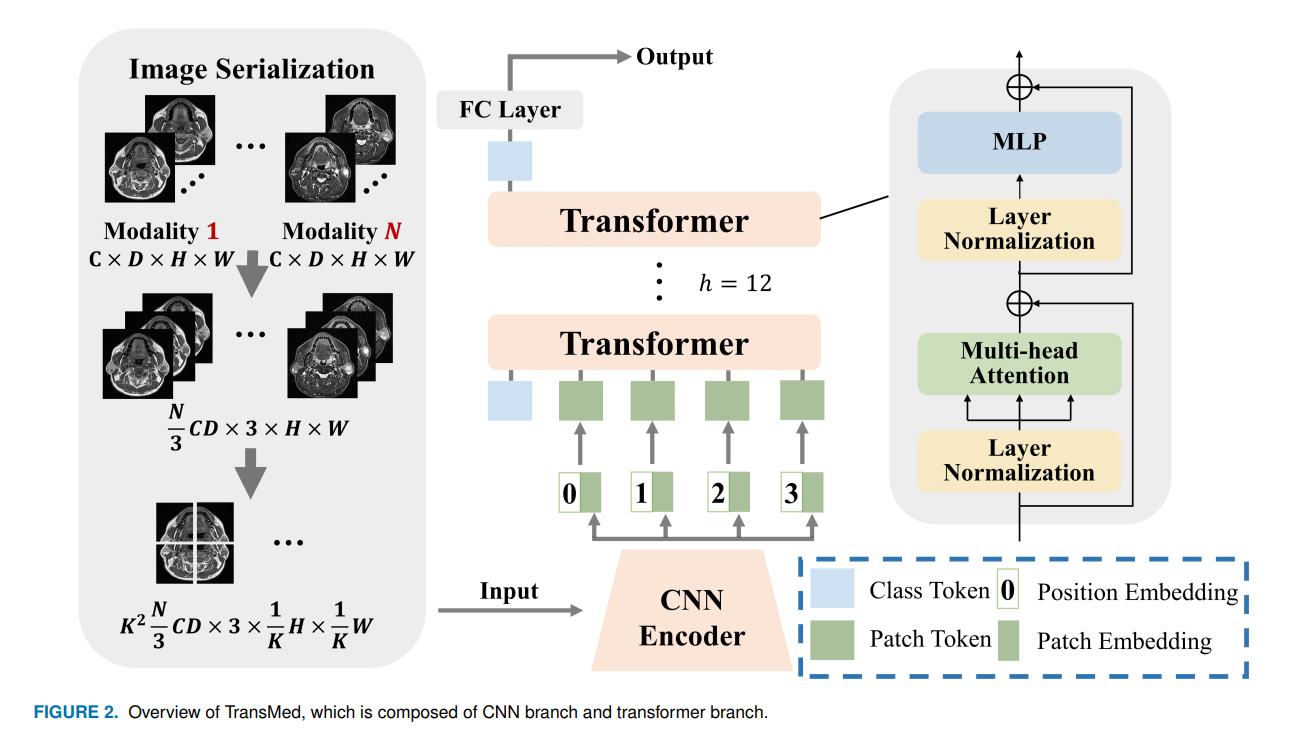
transformer聚集多模态特征
作者尽可能采取DeiT的实现。这样有意简单设置的优点在于减少其他trick的对模型性能的影响,并且直接显示了transformer带来的优点。此外,我们使用了拓展的DeiT模型和它的预训练权重。
transformer的重要组件包括自注意(SA),多头注意力(MSA),和多层感知器(MLP)。transformer的输入包括一系列embeddings和tokens。不同于DeiT,作者移除了线性映射层和distillation token。
-
self-Attention:SA是一个注意机制,它使用同一样本的其他部分来预测数据样本的其他部分。在计算视觉中,它与非局部网络有一些相似。SA有很多种形式,常见的transformer依赖放缩点乘的形式。
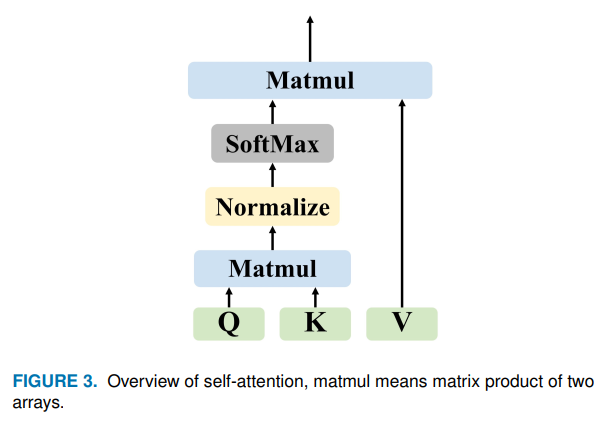
在SA层,输入向量首先被变换成3种不同向量:Query matrix Q, key matrix K和 value matrix V, 输出是value向量的权重和。每个value的权重由query和相对应的key点积决定。不同输入向量的注意力函数如下公式计算
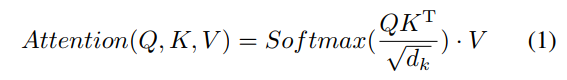
$d_k$是key向量k的维度。$\sqrt{d_k}$可以使梯度更加稳定的恰当的正则化
-
Multi-head Self-Attention: MSA是transformer的核心组件。正如下图所示,与SA的不同之处在于多头机制将输入分离成很多小部分,然后并行地计算每个输入地缩放点积,最终拼接所有地注意力输出来得到最终地结果。MSA的公式可以看成:

映射$W^Q_i$,$W^K_i$,$W^V_i$和$W^O$是可训练参数矩阵,h是transformer层的数量。MSA的优点在于它允许模型在不同表示子空间学习序列和定位信息。
-
Multi-Layer Perceptron:在本文中,MLP被添加到MSA层的顶层。MLP由被GeLU分离的线性层组成。MSA和MLP都有如残差一样的跳跃连接和层归一化。因此假定$t-1$层的表示为$x_{t-1}$, LN表示线性归一化,$t$层的输出可以写成
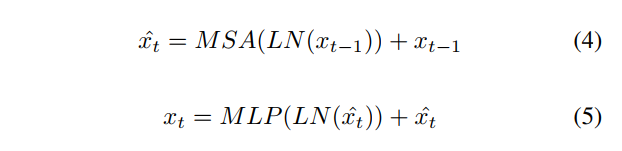
-
Embedding and Tokens:输入层包含5个embeddings和tokens,分别是patch embedding, position embedding, class embedding, patch token, class token。
patch embedding是每个patch的CNN的输出, class embedding是可训练向量。为了将patch的空域信息和位置信息编码到patch token, 作者使用了position embeddings和patch embeddings来保留信息。Class embedding没有可以添加的patch embedding, 因此class token和class embedding是相等的(相同的?)。假定输入是x,可训练向量是$W^c$,position embedding是$x_{po}$, patch token是$x_{pt}$,class token $x_{ct}$可以表示成如下形式
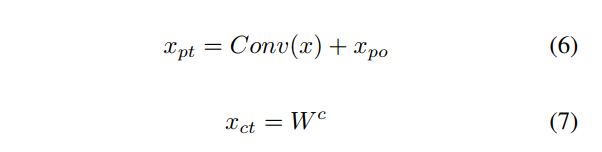
在transformer的输入层之前,class token与patch tokens系在一起,然后输入transformer 层并且从全连接层输出来预测类别。
TransMed
TransMed的结构如图一所示。与直接使用纯transformer作为编码器不同,TransMed采取了一个混合模型包括CNN和transformer,其中CNN被当作一个底层特征提取器来生成patch embedding。
给定一张多模态图像 $x\in R^{B \times M \times C \times D \times H \times W}$,其中空域分辨率为$H \times W$,深度为D(同一次检查的不同方向?),通道数为C,模态的数量为M(不同时期检查?),批大小为B。在输入到CNN编码器之前,有必要构建序列。首先,多模态图像的三个邻近D分离被叠加构建成3通道图像。然后, 每张图像被分割成$K \times K$。K的值越大意味着每个Patch就越小。作者评估了不同K值对模型性能的影响。最终,图像被编码成$(\frac{1}{3}BMCDK^2, 3, \frac{H}{K}, \frac{W}{K})$个patch。
在图像序列被构建以后,将其输入2D CNN中。最终2D CNN的全连接由线性投影层替代,将向量patch映射到潜在的嵌入空间。2D CNN从图像序列中提取底层特征,并将其编码成初级形式,输出的shape为$(B,\frac{1}{3}MCDK^2,P)$,其中p的大小被设置成自适应transformer的输入大小。
结果
数据集和预处理
数据集:344个患者的MRI的两个模态,ground truth标签由生物活检得到。将腮腺肿瘤分割成5个类别。
预处理:首先使用OTSU来提取原始图像中的前景部分。然后相同病人的不同模态图像被注册到前景区域提升一致性。然后重采样每张图像到(18,448,448)。因此,最终得到344张图像,由MRI TI 和T2的3D图像堆叠而成,尺寸是(36,488,448)。数据增强使用了随机翻转(50%概率)和随机噪声(0均值,0.1方差的高斯噪声)。
结果
- TransMed以更少量的参数和更低的计算代价获得了SOTA的性能.
- TransMed可以有效的建模模态之间的长期关系。
- 当K值很大时,性能很差。可能原因是图像块的尺寸过小损害了图像中的语义信息。
单词
- lesion 病灶
- parotid gland tumors 腮腺肿瘤
感悟:用上了最新的RTX3080,在医学图像分析领域率先使用了transformer结构,该文章如果投在顶刊上,估计拖个一年出来就不是第一个用transformer做的工作了。虽然ACCESS风评不好,但是行业首篇如果在以后能收货可观的引用量的话,其实期刊本身带来的负面影响将会降低。